Nell’articolo precedente abbiamo analizzato le tecniche di sequenziamento appartenente alla categoria nota come “sequenziamento di prima generazione“. Nel corso del tempo si sono sviluppate nuove tecniche che hanno reso più economico e di conseguenza più accessibile sequenziare frammenti di interesse. Di seguito parleremo del sequenziamento di seconda generazione o NGS (Next Generation Sequencing).
Caratteristiche generali Next generation sequencing
Il sequenziamento di seconda generazione utilizza un procedimento molto simile a quello del Sequenziamento Sanger tuttavia la principale differenza risiede nella quantità di campione che è possibile sequenziare. Mentre il metodo Sanger permette di sequenziare un frammento di DNA di modeste dimensioni (700 – 1000 paia di basi), attraverso l’utilizzo della tecnologia NGS si è in grado di sequenziare più frammenti di DNA simultaneamente. I sistemi di sequenziamento adoperati nella tecnologia di seconda generazione sono molteplici, ognuno di questi è sviluppato da un’azienda differente, e sono elencati qui di seguito:
- Roche 454 System;
- AB SOLiD System;
- Heliscope;
- Illumina GA/HiSeq System.
Ciononostante parleremo nello specifico del sequenziamento che prevede l’utilizzo del sistema Illumina che rappresenta una delle tecniche più utilizzate oggigiorno.
Funzionamento Next generation sequencing
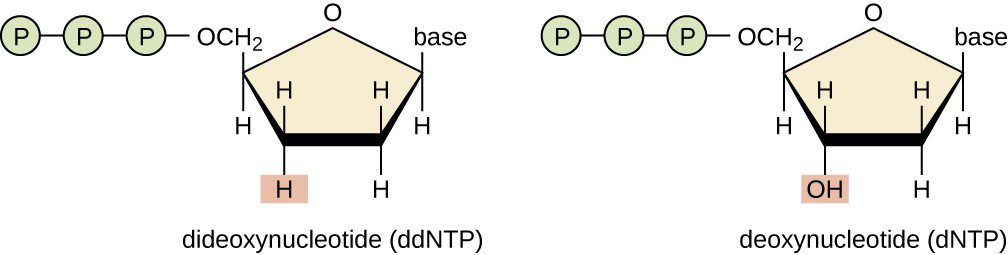
Il sequenziamento di seconda generazione, come il sequenziamento Sanger automatizzato, utilizza dideossiribonucleotidi con un fluorocromo differente per ogni dideossiribonucleotide (ddATP, ddTTP, ddGTP, ddCTP). (Figura 1)
La reazione di sequenziamento avviene su di una cella di flusso (flow cell) e richiede i seguenti componenti:
- Frammenti di DNA da sequenziare denaturati;
- Primer, sequenza oligonucleotidica, complementare alla regione fiancheggiante al frammento di DNA da sequenziare;
- Adattatori o “adapters“, necessari per l’adesione dei frammenti al supporto;
- DNA polimerasi ad alta processività;
- dNTP, deossiribonucleotidi trifosfati;
- ddNTP dideossiribonucleotide trifosfato.
Procedimento
Il sequenziamento di seconda generazione è possibile suddividerla in quattro fasi principali.
Frammentazione e amplificazione
La prima parte consiste nella frammentazione casuale dei campioni di DNA e il successivo legame con gli adattatori che si legano all’estremità 5′ e 3′ dei frammenti in modo che possano attaccarsi al supporto. Gli adattatori presentano tre regioni. La prima regione permette ai vari frammenti di interagire con gli oligonucleotidi presenti sul supporto mentre l’indice è una regione di 6 – 8 paia di basi che permette di distinguere i vari adattatori, infatti questa regione permette di distinguere diversi campioni e quindi di sequenziarne più di un campione. Infine una regione di legame per i premier di sequenziamento. I frammenti così preparati vanno a costituire la nostra libreria di sequenziamento. (Figura 2)
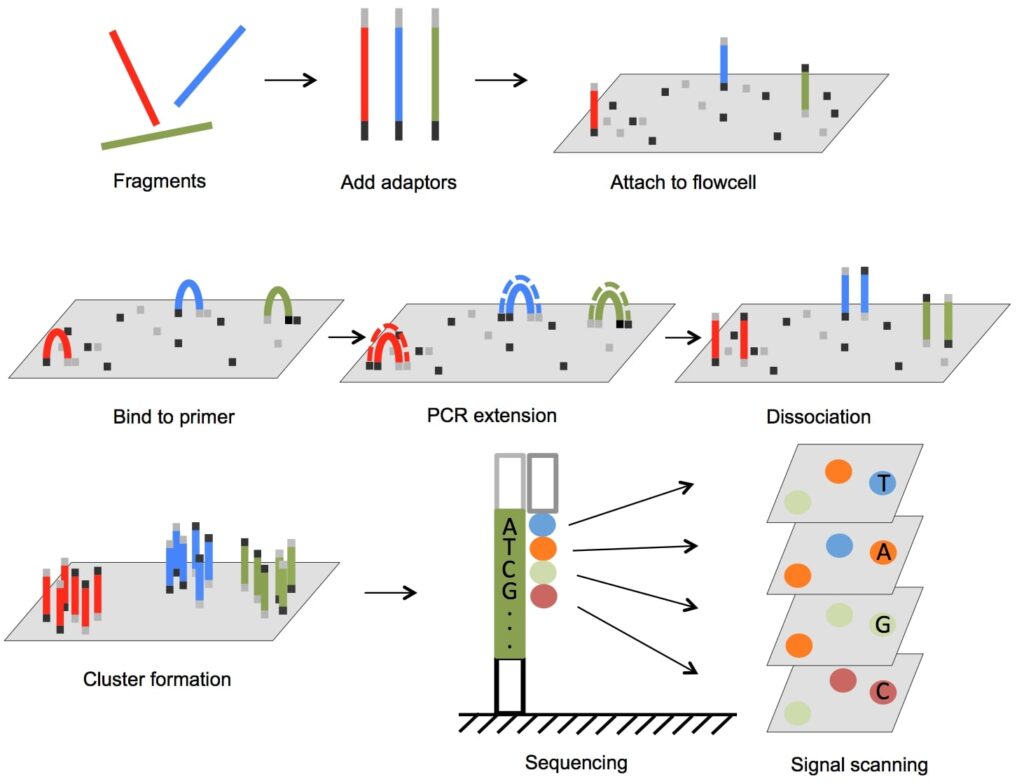
A seguire vengono aggiunti i dNTP, deossiribonucleotidi trifosfati, e la polimerasi che sono necessari per l’amplificazione; questo processo di amplificazione è noto come “bridge amplification” ovvero amplificazione a ponte. I frammenti, poi, si ibrideranno con gli oligonucleotidi presenti sul supporto e legheranno i primer adiacenti formando la tipica struttura a ponte. In seguito agisce la polimerasi che sintetizza il filamento opposto e tale processo viene ripetuto più volte. Alla fine per preparare i filamenti al sequenziamento si esegue la linearizzazione, dopo di ciò sarà possibile osservare la formazione di diversi clusters.
Sequenziamento e analisi dei dati
Il sequenziamento del sistema Illumina viene definito anche “sequenziamento mediante sintesi” (sequencing by synthesis), avviene attraverso l’uso di ddNTP differenti ognuno dei quali legato con un fluorocromo diverso, la polimerasi e un primer complementare ad una regione dell’adattatore. Quando uno di questi ddNTP viene incorporato nel filamento, l’emissione di fluorescenza viene rilevata da uno specifico detector dopodiché il software registra le diverse emissioni di fluorescenza e le converte nella sequenza nucleotidica corrispettiva. Il sequenziamento può avvenire partendo da una singola estremità (single-end reads) in 2 – 3 giorni mentre se avviene da entrambe le estremità (paired-end reads) il sequenziamento avverrà in 4 – 6 giorni. (Figura 2 )
I frammenti sequenziati (reads) dal sequenziamento sono milioni quindi questo richiede un’analisi molto elaborata per la ricostruzione della sequenza. I metodi di assemblaggio possono prevedere due procedure:
- Assemblaggio del genoma basato su sequenze presenti all’interno di software bioinformatici (Reference based genome assembly);
- Assemblaggio del genoma de novo (De novo genome assembly).
Limitazioni del sequenziamento di seconda generazione
Nonostante il sequenziamento di seconda generazione sia meno costoso del sequenziamento Sanger, questo permette il sequenziamento di piccoli frammenti (50 – 250 paia di basi), in più il tempo necessario per il sequenziamento dei frammenti può richiedere fino a sei giorni a seconda da dove inizia il sequenziamento del frammento.
Ismael Sanchez Polanco
Fonti
- “Understanding the Basics of NGS: From Mechanism to Variant Calling” Dale Muzzey, Eric A. Evans, Caroline Lieber. Curr Genet Med Rep (2015) 3:158–165
- https://emea.illumina.com
- https://www.illumina.com
I ddNTP interrompono la polimerasi (la quale ha bisogno di un gruppo – OH al 3′ per poter funzionare) e questo viene sfruttato nel Sanger, in lllumina si utilizzano dNTP modificati che presentano un terminatore reversibile. Se si utilizzassero ddNTP non si potrebbe parlare di sequenziamento mediante sintesi