qPCR e dPCR
La real time PCR o reazione quantitativa a catena dell’enzima DNA polimerasi è una tecnica ampiamente utilizzata in laboratori (ricerca clinica, ricerca veterinaria, ricerca agraria). E’ usata per svolgere attività di diagnosi molecolare su qualsiasi matrice per la rilevazione di una specifica sequenza di DNA contenuta in uno o più organismi di interesse. La quantificazione in tempo reale è l’aspetto che ha svoltato la normale routine da PCR tradizionale ed è ancora preferita su recenti versioni della reazione, quale la digital PCR. Quest’ultima è più recente e risale al 2016; mostra una applicazione della reazione PCR in formato maxi, posta sotto lente d’ingrandimento per la sperimentazione in routine.
qPCR
La PCR quantitativa permette la rilevazione e quantificazione di una specifica sezione del DNA. Le applicazioni comprendono:
- tipizzazione di agenti microbici contaminanti e patogeni;
- identificazione di ceppi/isolati e/o di profili resistenza antimicrobica e produzione tossine.
Può essere applicata sia per molecole a singolo filamento RNA sia per DNA. Infatti, l’inserimento nella reazione dell’enzima retro-trascrittasi permette di originare DNA dall’RNA in modo da essere inserita nella reazione a protocollo standard DNA. Lo sviluppo della reazione qPCR consta di una fase pre-amplificazione, di una seconda di amplificazione e di una terza post-amplificazione.
qPCR: prima fase (preparazione)
La prima fase, solitamente si effettua sotto cappa in sterilità e permette di ottenere una provetta contenente reagenti chimici stabilizzanti e performanti, DNA polimerasi del Thermus acquaticus, il DNA estratto di un organismo e/o microrganismo, sequenze oligonucleotidiche innesco del DNA target, sonde fluorescenti complementari al target, oligonucleotidi liberi ed acqua. Ulteriore lavoro viene svolto al fine dell’ottenimento di campioni privi di DNA, alcuni per la diluizione seriale del DNA target ed altri controlli positivi.
qPCR: seconda fase (amplificazione)
La seconda fase, consiste nel settare il software manualmente, di inserire la piastra dei campioni nel ciclatore e di avviare la reazione che in media dura 1 ora e 30 minuti e comprende massimo 40 cicli sebbene estendibili a 50 e più che ripetono per ogni una fase di denaturazione, una di allineamento ed una di sintesi.

qPCR: terza fase (biostatistica)
Al termine, si utilizza il software per ottenere una curva di calibrazione che è usata per identificare, in ordine logaritmico trasformabile, la quantità di DNA target, se presente, in ogni campione. A questa retta, si aggiungono analisi di efficienza della reazione, la specificità-selettività degli inneschi per il target, inclusività ISO 16140 per 50 e più ceppi, sensibilità con il campione bianco (privo di DNA), limite di rilevazione (LOD) e di quantificazione (LOQ), accuratezza del campione riferimento sul campione in analisi.
La reazione di amplificazione risulta corretta e priva di falsi-positivi ed altri errori se ogni parametro è ben studiato antecedentemente, come ad esempio il disegno specifico degli inneschi. Infatti, essi sono disponibili se è già stato sequenziato il genoma dell’organismo o almeno una parte di interesse. La reazione è veloce e ad alto flusso e permette la quantificazione in diverse matrici ed anche simultaneamente per diversi target DNA provenienti da due o più organismi. Gli errori talvolta, derivano da difetti dell’apparecchiatura e/o dalla lavorazione manuale dell’operatore e/o bassa qualità reagenti ed estratti molecolari.
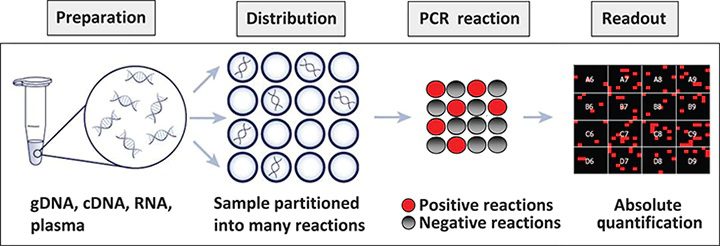
dPCR
La digital PCR è la più innovativa versione della reazione. Consiste nella distribuzione casuale del DNA in eguali partizioni e nel successivo avvio di un elevato numero di reazioni. Nasce dall’esigenza di trovare una tecnica molecolare in grado di identificare il singolo nucleotide differente (SNP) di una singola sequenza in migliaia di molecole. Infatti, è ancora usata maggiormente come tecnica di diagnosi di mutazioni rare. Permette una più semplice quantificazione del DNA con lettura autonoma senza dover allestire una retta di taratura.
La tecnica mostra elevata sensibilità e riproducibilità, ma ancora una procedura complessa e/o non ben descritta e nota. Con questa reazione aumentano i tempi pre- e post-amplificazione, il che fa preferire ancora la qPCR per la diagnosi in routine. La quantificazione nella dPCR è talmente precisa da riuscire a rilevare concentrazioni molto basse che in qPCR non si riuscirebbe altrimenti ad identificare se non con ripetizione e arricchimenti.
I quantificati a bassa concentrazione devono altresì essere confermati con la ripetizione della reazione: aspetto che può essere gestito all’interno della stessa se dovesse aumentare il numero di campionamenti disponibili.
Risulta importante misurare la variabilità dei volumi; infatti una bassa variabilità di questi e una bassa concentrazione del campione corrispondono ad un effetto non rilevante e trascurabile da falso positivo.
Riferimenti
- A Basic Guide to Real Time PCR in Microbial Diagnostics: Definitions, Parameters, and Everything; Review ARTICLE Front. Microbiol., 02 February 2017 | https://doi.org/10.3389/fmicb.2017.00108, Petr Kralik and Matteo Ricchi, Department of Food and Feed Safety, Veterinary Research Institute, Brno, Czechia and Istituto Zooprofilattico Sperimentale della Lombardia e dell’Emilia Romagna “Bruno Ubertini,” National Reference Centre for Paratuberculosis, Piacenza, Italy.
- Considerations for Digital PCR as an Accurate Molecular Diagnostic Tool; Clinical Chemistry 61:1 79–88 (2015), Jim F. Huggett, Simon Cowen and Carole A. Foy, LGC, Teddington, UK; Research Department of Infection, Division of Infection and Immunity, UCL, London, UK.
- Digital PCR: a new technology for diagnosis of parasitic infections, E. Pomari, C. Piubelli, F. Perandin, Z.Bisoffi, https://doi.org/10.1016/j.cmi.2019.06.009, Clinical Microbiology and Infection, Volume 25, Issue 12, December 2019, Pages 1510-1516
- https://eurofinsgenomics.eu/en/qpcr/
- https://www.thermofisher.com/it/en/home/life-science/pcr/digital-pcr.html.html