Principio dell’estrazione del DNA
Il DNA è una molecola da cui possono essere estrapolate numerose informazioni mediante lo sviluppo e l’applicazione di varie tecniche molecolari come la PCR, l’elettroforesi, il Southern Blot e diverse tecniche di sequenziamento. Tale molecola è preziosissima per studi di ricerca clinica, nelle produzioni animali e nelle analisi forensi. Prima di poter analizzare l’acido nucleico però, è necessario estrarlo e purificarlo al fine ottenere dei risultati ottimali ed attendibili.

Materiale biologico di partenza per l’estrazione del DNA
Il DNA può essere estratto da diverse tipologie di materiali; per rilevare l’acido nucleico, i campioni prelevati devono essere costituiti da materiale biologico o quantomeno devono preservarne delle tracce.
Alcune matrici di partenza possono essere:
- Liquidi corporei: sangue, saliva, urine, liquido amniotico, liquido seminale;
- Feci;
- Cellule provenienti da colture cellulari;
- Campioni tissutali: freschi, prelevati mediante biopsia, congelati oppure conservati mediante fissazione in formalina ed immersione in paraffina;
- Residui biologici di feci, urine, saliva, peli o capelli;
- Batteri e parassiti.
La matrice di partenza da cui dovrà essere estratto il DNA deve essere opportunamente conservata a basse temperature. Se il campione estratto viene processato in tempi brevi è possibile conservarlo a +4°C per 24-48h al massimo. In caso di tempi di elaborazione più lunghi il campione deve essere opportunamente conservato ad una temperatura compresa tra -20°C e -80°C. Il DNA è una molecola facilmente degradabile per questo necessita di temperature tanto basse.
Metodo dell’estrazione del DNA
Esistono varie tecniche di estrazione del DNA che spaziano dalla classica estrazione mediante fenolo-cloroformio fino all’utilizzo di svariati kit commerciali che velocizzano e facilitano senz’altro la procedura. Al di là della tecnica applicata, l’obiettivo principale del procedimento di estrazione è ottenere un buon quantitativo di acido nucleico caratterizzato da un elevato grado di purezza, quindi privo di contaminazioni, al fine di agevolare le successive tecniche di studio e analisi.
Per rendere tangibile la tecnica con gli obiettivi preposti è necessario focalizzarsi sul corretto svolgimento di alcuni punti generali:
- La lisi cellulare con un corretto rilascio del contenuto intracellulare e nucleare.
- La separazione degli acidi nucleici dal resto delle molecole, quali lipidi, proteine e carboidrati.
- L’eliminazione e la degradazione dell’RNA mediante l’utilizzo di RNasi.
- Il recupero degli acidi nucleici mediante precipitazione alcolica e purificazione.
- La quantificazione e la valutazione del grado di purezza dell’acido nucleico estratto.
Fase di lisi
La fase di lisi deve essere molto decisa e potente al fine di degradare il tessuto o le cellule di partenza in modo da versare esternamente contenuto intracellulare. Tuttavia, questa fase non deve causare la frammentazione degli acidi nucleici.
La lisi può essere di varie tipologie:
- meccanica: consiste nell’utilizzo di pestello e mortaio, sfere di vetro agitate con strumenti come il TissueLyser, miscelatori vortex, o mediante l’applicazione dell’ultrasonicazione o dell’elettroporazione.
- chimica: si serve di detergenti contenenti dei sali caotropici che provocano la rapida denaturazione delle proteine e destabilizzano le interazioni idrofobiche. In questo modo si verifica la rottura delle membrane lipidiche con conseguente riversamento all’esterno del contenuto cellulare.
- enzimatica: adopera enzimi litici, sfruttati soprattutto per la rottura della parete cellulare dei batteri, come ad esempio il lisozima.
Estrazione di DNA mediante Fenolo-Cloroformio
L’estrazione di DNA mediante Fenolo-Cloroformio è una metodica definita in “fase liquida” poiché si svolge solamente tramite l’utilizzo di soluzioni liquide e non prevede alcuna fase solida.
In seguito alla fase di lisi è di fondamentale importanza digerire il quantitativo proteico presente nel campione. Questo passaggio evita la degradazione del DNA da parte di specifiche proteine presenti nel contenuto cellulare. La digestione inoltre permette di ottenere un grado di purezza cospicuo dell’acido nucleico. Tale fase avviene mediante l’aggiunta di enzimi proteolitici come pronasi o proteinasi K.
- La pronasi è una miscela di 4 enzimi proteolitici, due endopeptidasi e due esopeptidasi.
- La proteinasi K è un’endopeptidasi di origine fungina capace di idrolizzare la cheratina.
Successivamente si aggiungono le RNasi per eliminare l’RNA presente nel campione.
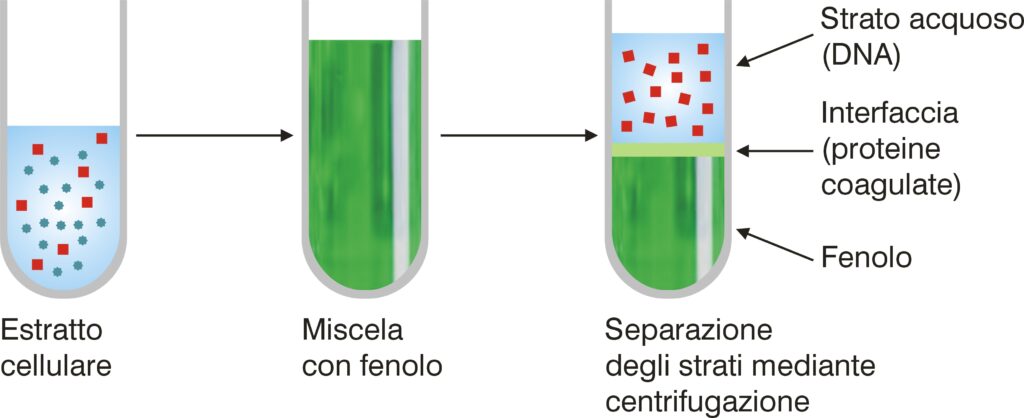
Un altro metodo per allontanare le proteine associate agli acidi nucleici è l’estrazione con fenolo. Alla soluzione acquosa contenente DNA viene aggiunta una miscela, immiscibile in acqua, di fenolo, cloroformio e alcol isoamilico.
Si procede centrifugando la soluzione, ottenendo al termine tre fasi distinte:
- la fase superiore, acquosa, contiene gli acidi nucleici;
- l’interfase è costituita da proteine denaturate e in parte dissolte nel fenolo;
- la fase inferiore, detta anche fenolica, contiene una componente organica formata da lipidi e proteine ricche di amminoacidi idrofobici.
In seguito, si procede con la precipitazione alcolica del DNA in etanolo assoluto in presenza di sali. Tale precipitazione, in presenza di concentrazioni relativamente alte di ioni monovalenti, è indispensabile per concentrare la soluzione di DNA ed eliminare i residui di fenolo e cloroformio che possono interferire nelle successive analisi molecolari.
L’etanolo modifica strutturalmente gli acidi nucleici che si aggregano e precipitano per poi essere recuperati.
Estrazione mediante kit commerciali
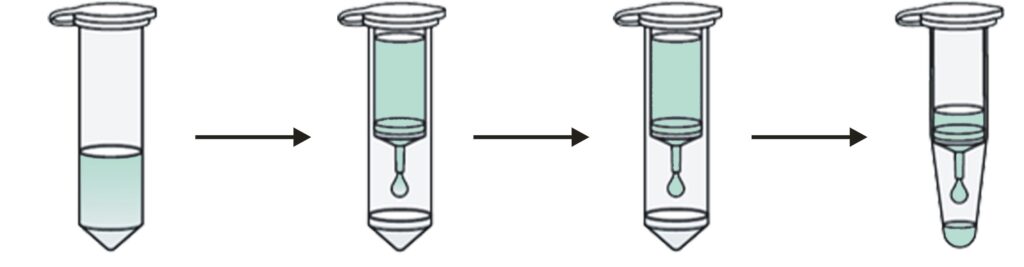
Esistono anche altri metodi, molto comuni e standardizzati, detti “in fase solida” in quanto utilizzano un supporto solido che trattiene l’acido nucleico. In commercio esistono diversi kit che utilizzano il metodo in fase solida e i supporti utilizzati sono delle membrane di silice.
Il principio base del kit è rappresentato da una membrana di silice, contenuta in una colonnina, in grado di legare e trattenere il DNA. Il legame con la silice della membrana è favorito dalla presenza di sali caotropici nel tampone di lisi. Questi sali destabilizzano le proteine e interrompono i legami tra acidi nucleici ed acqua, fornendo quindi condizioni ottimali per il legame del DNA con la membrana.
I residui di RNA sono successivamente rimossi mediante un trattamento con RNasi.
La membrana di silice viene lavata solitamente con etanolo al 70% in modo da allontanare i contaminanti. Infine si procede con la fase di eluizione utilizzando acqua o tamponi a bassa concentrazione di sali che permettono il distacco dell’acido nucleico dalla membrana cambiando il ph della soluzione. Anche se il DNA è legato alla membrana ed è lavato con etanolo necessita ugualmente di ulteriori lavaggi per essere pulito al meglio dagli ultimi residui di sali e proteine in modo da ottenere un elevato grado di purezza.
Utilizzando questo metodo si ottiene sicuramente un acido nucleico molto puro, ma la membrana di silice tratterrà una piccola quantità della molecola per cui la resa è inferiore rispetto al metodo in fase liquida.
I metodi in fase solida sono rapidi, semplici, standardizzabili ed è possibile riprodurli in sistemi automatizzati.
Estrazione mediante sfere magnetiche
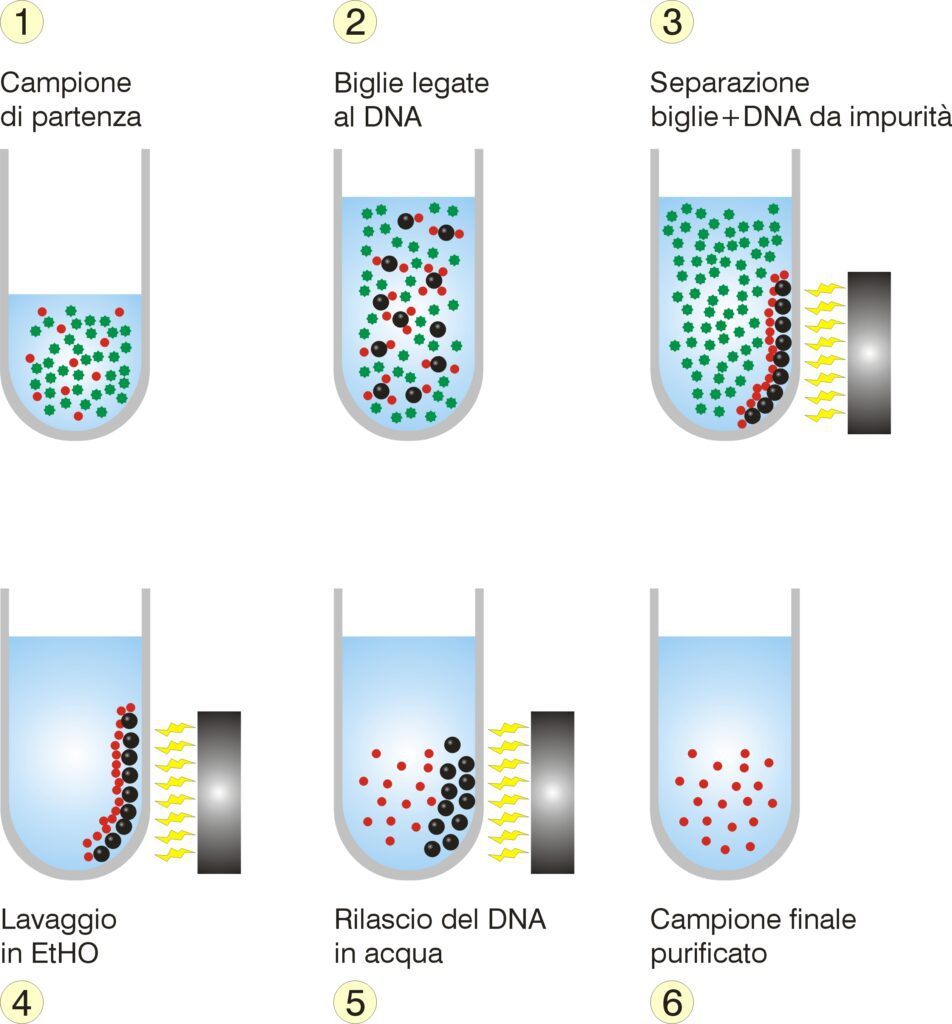
Alcuni metodi commerciali utilizzano particelle magnetiche che agiscono come fase solida attorno alla quale si lega l’acido nucleico, quindi non necessitano di alcun passaggio di centrifugazione e filtrazione. Il sistema di estrazione automatizzato che utilizza sfere magnetiche prevede, dopo la fase di lisi e la digestione con proteinasi K, il trasferimento della sospensione ottenuta in colonnine contenenti delle piccole sfere magnetiche rivestite con resina di silice. La silice permette il legame dell’acido nucleico alle sfere.
In seguito si attiva una piastra magnetica che attira e trattiene le biglie caricate con il DNA sul fondo della colonna. Questo passaggio permette un’agevole aspirazione del surnatante contenente i contaminanti da scartare. Successivamente, le biglie vengono sottoposte a vari lavaggi rapidi per eliminare ulteriori contaminanti e sali. Infine, il magnete viene allontanato e l’acido nucleico viene eluito con acqua o soluzioni tamponate (tris-EDTA), per essere purificato e separato dalle sfere che restano all’interno della colonnina.
Risultati attesi
La quantificazione del DNA viene comunemente eseguita per determinare la concentrazione dell’acido nucleico presente in soluzione ed il suo grado di purezza. Generalmente, per eseguire tale analisi viene sfruttata una quantificazione spettrofotometrica all’UV. L’analisi spettrofotometrica si basa sul grado di assorbanza dell’acido nucleico per valutarne il quantitativo.
Il campione viene riposto all’interno di una cuvetta, che sarà successivamente esposta ad una luce UV di lunghezza d’onda pari a 260 nm, ovvero il livello di assorbanza del DNA. Più luce viene assorbita dal campione, maggiore sarà la concentrazione dell’acido nucleico. Solitamente la presenza di un buon quantitativo di DNA viene rappresentata dal macchinario attraverso uno spettro di assorbimento dall’andamento gaussiano. Parallelamente alla quantificazione dell’acido nucleico, viene valutato anche il grado di contaminanti presenti nel campione.
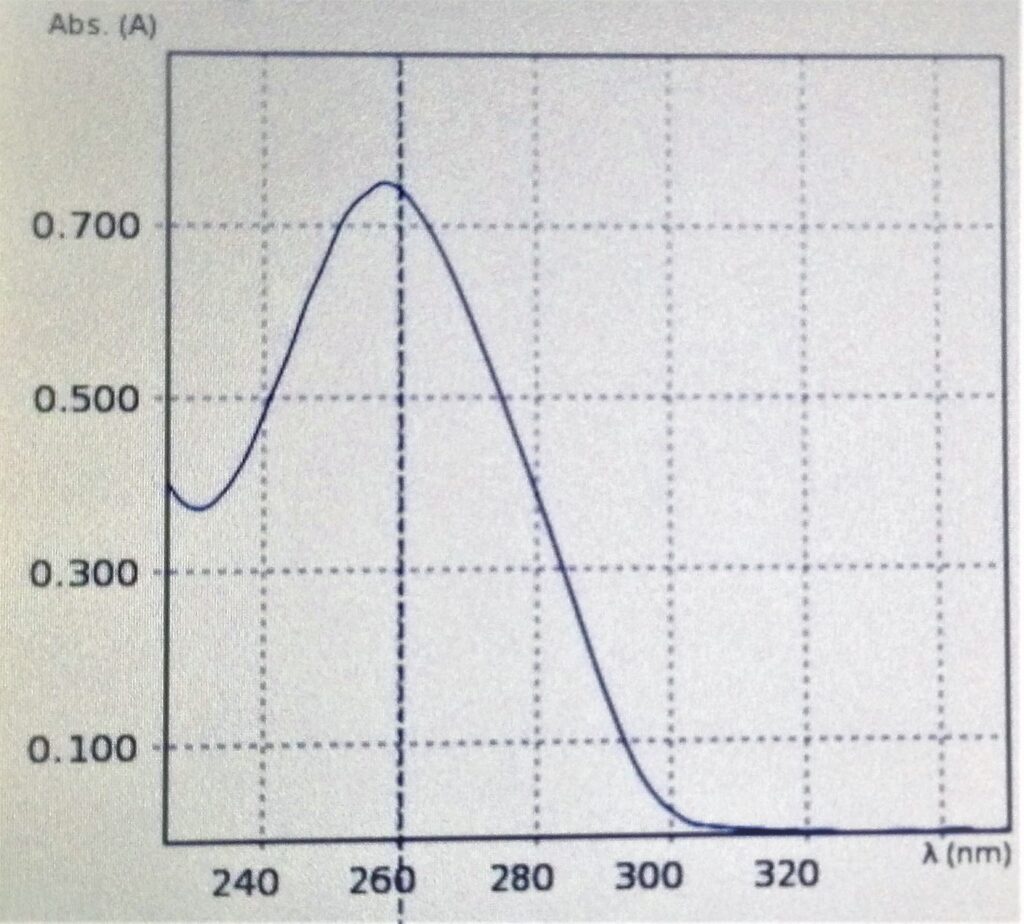
Limitazioni del test
La precisione del metodo estrattivo è sicuramente influenzata dalla qualità e dalla quantità del campione di partenza.
- Tessuti molto freschi sono soggetti a degradazione, quindi è necessario eseguire l’estrazione in tempi brevi. Il congelamento del tessuto risolve questa problematica, ma cicli di congelamento/scongelamento dell’intero campione tissutale sono da evitare poiché causano comunque la degradazione dell’acido nucleico. È preferibile estrarre l’acido nucleico, conservarlo nella sua purezza e scongelarlo solo al momento del bisogno evitando così la sua frammentazione.
- Tessuti conservati in fissativi si preservano per lunghi periodi. Tuttavia, questo tipo di conservazione danneggia la quantità e la qualità del DNA in quanto ne causa la frammentazione. La formalina è il fissativo per eccellenza, però frammenta il DNA. Un altro fissativo comunemente usato è l’alcol che, pur essendo ottimo per fissare l’acido nucleico, provoca artefatti cellulari a carico delle proteine. Quindi, tale componente fissativa spesso non è utilizzata per la conservazione di resezioni o biopsie per cui è richiesta anche un’indagine morfologica.
Altri tipi di limitazioni sono riconducibili alla metodica di estrazione applicata:
- I kit commerciali utilizzati per l’estrazione e la purificazione del DNA necessitano di numerosi trasferimenti dei campioni. Tale procedura favorisce la perdita del materiale genetico. Inoltre, vi è un maggiore rischio di contaminazioni, è una tecnica che richiede tempi lunghi ed il numero di campioni processati risulta limitato. Tuttavia, permette di ottenere un acido nucleico altamente purificato con una resa accettabile nel range di 100-150 ng/ul e preserva la lunghezza della molecola di DNA senza frammentarla.
- L’estrazione mediante biglie magnetiche è svantaggiosa perché molto costosa, inoltre garantisce una bassa resa pari a 5-20 ng/ul e il DNA risulta piuttosto frammentato. Tuttavia è una procedura veloce e automizzata, il DNA risulta altamente purificato con una bassa probabilità di contaminazione.
Quality control
Il controllo della qualità dell’estrazione e della purificazione del DNA si verifica mediante l’analisi spettrofotometrica. I parametri 260/280 e 260/230 permettono di capire se il materiale estratto è molto puro oppure risulta contaminato.
- Il rapporto 260/280 valuta il grado di contaminazione della matrice da parte di componenti proteiche, poiché il grado di assorbanza degli amminoacidi è 280 nm. Acidi nucleici di buona qualità solitamente producono un rapporto di 260/280 pari a 1,8. Livelli molto distanti da questi parametri possono indicare la presenza di vari contaminanti come fenolo, proteine e altri composti che assorbono in un range molto vicino a 280 nm.
- Il rapporto 260/230 invece, permette di identificare il grado di contaminazione del campione con ioni fenolato e altri composti organici. Il rapporto delle assorbanze a 260/230 deve essere compreso tra 2-2,2. Se i valori ottenuti sono inferiori a tale range è molto probabile che ci sia la presenza di contaminanti e che la tecnica di estrazione debba essere ottimizzata.
ciao sono un medico e biologo posso chiederti un informazione, conosci le varie modalità di preparazione delle sfere per la metodica magnetica?
Se puo ti ringrazio