La tecnica Shotgun metagenomic implicata nello studio del microbiota degli alimenti
La tecnologia Shotgun metagenomic, rappresenta il “golden standard” delle metodologie per lo studio dei vari consorzi microbici in natura. Tale strategia si applica allo studio di vari microbioti, da quello alimentare, passando per il microbiota intestinale.
Il sequenziamento Shotgun, va ad identificare in modo olistico tutto il materiale gentico (in questo caso RNA) che compone una matrice complessa.
Possiamo definire quindi la procedura Shotgun metagenomic, come un approccio molto preciso dato che non si basa su una pre-amplificazione del materiale genetico (evitando bias dovuti alla PCR). Essendo un sistema cultura indipendente, si possono avere un gran quantitativo di dati in poco tempo, questa peculiarità conferisce a tale metodologia lo status di “tecnica high throughput”.
Questi vantaggi permettono di:
- espandere la conoscenza dei genomi batterici.
- Studiare i fenomeni di cross talk metabolici.
- Studiare l’induzione di fattori virulenza/resistenza.
- Premettere un miglior controllo e caratterizzazione del prodotto.
Non dobbiamo tralasciare che analizzando solo la parte trascritta, andiamo a visionare i geni ed i microrganismi biologicamente attivi in un dato alimento.
Cenni di funzionamento della tecnologia Shotgun metagenomics
La tecnologia Shotgun metagenomics, si basa sul sequenziamento di seconda generazione (con un classico protocollo “Next-gen”).
Essendo una tecnica “high throughput”, e quindi producendo tanti output di sequenziamento (reads) in poco tempo; per mezzo dell’ausilio di genomi di riferimento attraverso un “matching” in banche dati note, si riescono a ricostruire i genomi che compongono il microbiota di una data matrice alimentare.
Tutto questo è possibile attraverso software bioinformatici.
Quindi, tale strategia, si prefigge lo scopo di studiare il microbioma tipico, con il fine di svolgere un’analisi tassonomica delle specie che lo compongono.
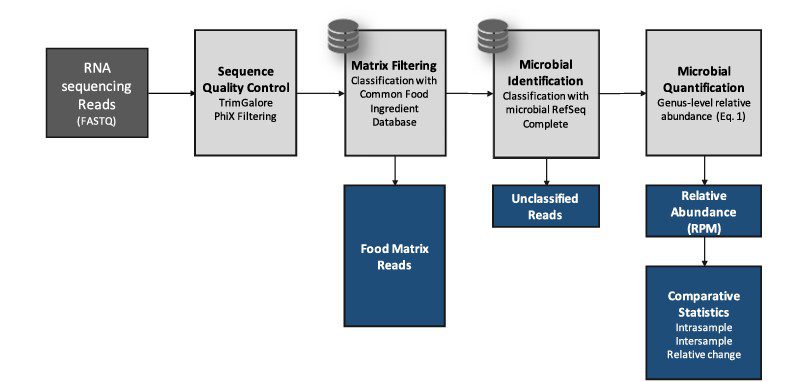
Si deve considerare, che in codesta guida, più di metagenomica, ci si riferisce alla metatrascrittomica dato che si considera il sequenziamento dell’RNA totale.
L’applicazione della tecnologia Shotgun metagenomic nella rintracciabilità alimentare
Recenti studi condotti in condizioni controllate, hanno dimostrato come un’alterazione nell’ingredientistica di un particolare alimento, possa modularne il microbiota tipico.
La modulazione si ha, per l’effetto prebiotico giocato dai componenti di una particolare matrice alimentare, sui consorzi microbici che la compongono. Quindi il mutamento di un ingrediente è strettamente correlato, alla modifica delle abbondanze relative delle specie batteriche che costituiscono il microbiota tipico della matrice stessa.
In tutto questo la tecnologia Shotgun metagenomic, ci viene in aiuto per caratterizzare e controllare un dato alimento. Ad esempio, se una particolare matrice, è caratterizzata da ingredienti ben specifici, che promuovono la selezione di consorzi microbici tipici; un eventuale alterazione nella sua composizione comprometterebbe le abbondanze delle varie specie microbiche. Effettuando un’analisi Shotgun, attraverso il calcolo delle abbondanze relative tipiche dei microorganismi presenti in un alimento (valore espresso come α-diversity), si possono rilevare eventuali alterazioni delle composizioni batteriche, dovute ad errori di processo o per eventuali frodi alimentari.
Non solo alterazioni nella composizione degli ingredienti, ma per mezzo della tecnologia Shotgun metagenomic, studiando indirettamente il microbiota tipico di una matrice alimentare, prossimo capire se essa e contaminata da patogeni o meno.
Un esempio attuale: la contaminazione da parte di Salmonella spp.
La salmonellosi è una tossinfezione alimentare molto temuta, esistono migliaia di sierotipi del patogeno Salmonella spp. che ne rendono difficile l’individuazione e la diagnosi.
Sempre da studi condotti in vitro (Kristen L. Beck et.al), è emerso che un eventuale microrganismo alieno (in questo caso anche patogeno), modula il microbiota tipico di un alimento, attraverso rapporti di cross talk e feeding.
Alla luce di ciò, con la tecnologia Shotgun metagenomic; attraverso lo studio indiretto delle abbondanze relative tipiche del microbiota di un alimento, possiamo capire se esso è stato contaminato e di quale patogeno si tratta. Sempre riferendoci a Salmonella spp, la sua presenza in certi alimenti cambia le concentrazione di certe specie batteriche, che possono essere usate come biomarker per l’identificazione del patogeno. Così facendo si può evitare di usare una metodica complessa come il sequenziamento Shoutgun (che però è servito a rilevare i marker microbici), sostituendolo con tecniche più fruibili come una PCR specie specifica.
Il sequenziamento dell’RNA totale implicato nel miglioramento dei processi aziendali
La tecnologia Shotgun metagenomic, può essere sfruttata dalle aziende alimentari per controllare il processo produttivo, garantendo una standardizzazione dei paramenti di qualità.
Attraverso il calcolo della β-diversity, cioè la differenza intraspecifica a livello di diversità microbica fra i vari lotti confezionati, il produttore può mettere a punto strategie di controllo mirate.
Codesto approccio, promuoverebbe un miglioramento del processo produttivo, garantendo standard qualitativi sempre più elevati.
Conclusioni e considerazioni finali
Il sequenziamento Shotgun metagenomic, può essere un valido strumento diagnostico in campo di sanità pubblica, garantendo una rintracciabilità veloce, arginando eventuali focolai di tossinfezioni alimentari.
Tutto questo è possibile solo se si hanno a monte, banche dati genomiche ricche d’informazioni; altrimenti il “matching” fra i dati di sequenziamento ottenuti e quelli presenti in libreria non è significativo, e ciò indurrebbe ad errori di interpretazione.
Per quando riguarda l’utilizzo della tecnologia Shotgun metagenomic nelle aziende alimentari, c’è da dire che i costi d’utilizzo di una metodica così avanzata e le competenze necessarie per la gestione della stessa, comporterebbe alle imprese che la utilizzano, un forte incremento degli “Opex” (OPerational EXpenditure).
Fatto sta, che investire in tecnologie innovative come questa del sequenziamento dell’RNA totale, darebbe un vantaggio competitivo alle imprese che la utilizzano, migliorando anche il rapporto di fiducia produttore-acquirente.
Fonti
- https://journals.asm.org/doi/full/10.1128/aem.02120-17
- https://doi.org/10.1038/s41538-020-00083-y
- https://doi.org/10.1007/978-3-642-35161-7_2
Crediti immagini
- Immagine in evidenza:https://hulab.ucf.edu/research/projects/metagenomics/Metagenomics.html
- Figura 1: https://doi.org/10.1038/s41538-020-00083-y