Per labeling si intendono quelle tecniche che permettono di marcare molecole di interesse quali acidi nucleici, proteine o anticorpi. La marcatura è fondamentale per rilevare, isolare e analizzare le molecole di interesse.
Le sonde genetiche
Le sonde (probes) sono filamenti a singola elica di DNA o RNA in grado di riconoscere una sequenza nucleotidica specifica all’interno di un campione. Vengono utilizzate per cercare un determinato gene o una sequenza di DNA. Il riconoscimento avviene in seguito a una reazione di ibridazione tra la sonda e il bersaglio: le due molecole devono essere complementari per legarsi. In condizioni ottimali di alta selettività una sonda dovrebbe essere capace di individuare il cambio di una coppia di basi in una sequenza nucleotidica. Affinché l’ibridazione venga rilevata, è necessario che la sonda venga marcata.
Le sonde molecolari si possono sintetizzare nei seguenti modi:
- Clonaggio all’interno di vettori molecolari
- A partire dall’mRNA di interesse, si sfrutta la capacità della trascrittasi inversa per generare una molecola di cDNA
- Sfruttando la PCR
- Per sintesi chimica
In base allo scopo dell’esperimento, si possono usare sonde corte o lunghe. Gli oligonucleotidi corti sono formati da 10-50 basi. Usare sonde brevi ha i suoi vantaggi: sono molecole stabili, più facili da ottenere e ibridizzano più rapidamente (<30 min). Lo svantaggio delle sonde corte sta nel fatto che la marcatura e la sensibilità del labeling è minore. Invece, le sonde lunghe sono composte da centinaia di basi.
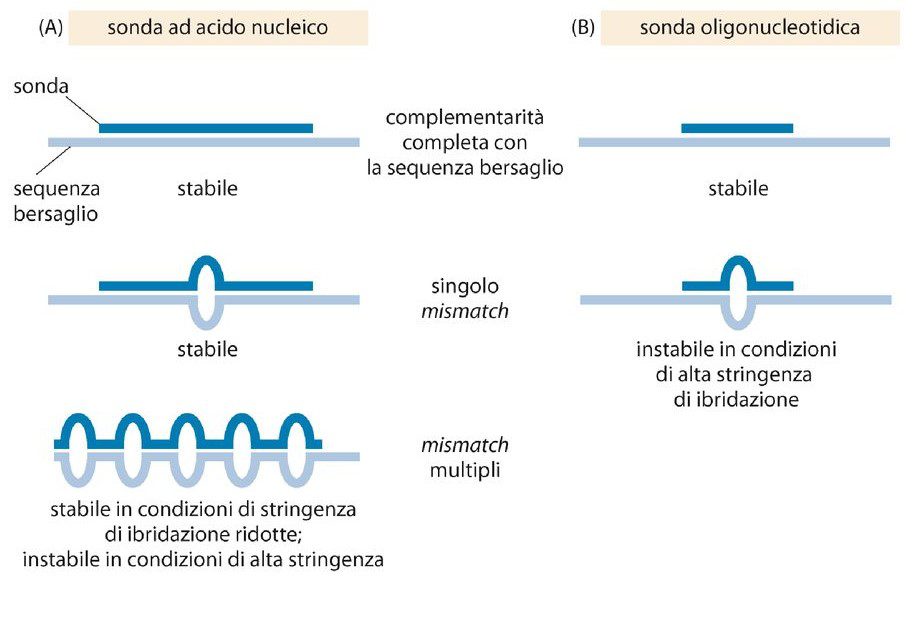
Le condizioni di stringenza
A seconda delle condizioni, la complementarietà tra la sequenza target e quella della sonda può non essere assoluta. Condizioni di alta stringenza si ottengono ricorrendo ad alte temperature, bassa concentrazione salina e presenza di agenti denaturanti. Tali condizioni prevengono gli appaiamenti imperfetti, per cui è necessaria un’elevata percentuale di omologia tra la sonda e la molecola target. Condizioni di bassa stringenza, invece, permettono l’utilizzo di sonde con una percentuale di omologia minore tra la sonda e la sequenza del target. Quest’ultima condizione è fondamentale qualora si vogliano studiare i geni omologhi presenti in specie diverse. Poiché la sequenza della sonda non è identica a quella del target, si deve garantire la formazione di una doppia elica stabile anche se l’appaiamento è imperfetto.
Labeling delle sonde
Le sonde genetiche devono essere marcate per permettere la rilevazione dell’ibridazione: a tal proposito, si possono impiegare isotopi radioattivi o molecole non radioattive. I sistemi di labeling radioattivi hanno maggior sensibilità rispetto all’impiego di sonde non radioattive. D’altra parte, l’utilizzo di tali traccianti richiede luoghi specializzati per la loro preparazione e il loro uso ed appropriati metodi di smaltimento. Inoltre, il laboratorio necessita un permesso specifico per il loro utilizzo e l’operatore deve ricevere un appropriato addestramento. Infine, ricordiamo che l’uso di questi traccianti, essendo radioattivi, sono pericolosi per la salute dell’operatore. Esistono diverse tecniche per marcare le sonde limitatamente alle estremità 3’OH o 5’P, oppure si può marcare l’intera molecola. La marcatura al livello delle estremità è particolarmente utile per i saggi che studiano le interazioni acido nucleico-proteina per evitare l’ingombro sterico.
Fosforilazione all’estremità 5′
Per marcare una sonda all’estremità 5’P, come prima cosa si fa interagire la molecola con una fosfatasi alcalina. La fosfatasi alcalina è un enzima che catalizza una reazione di defosforilazione della sonda all’estremità 5’P libera generando un gruppo alcolico. A questo punto interviene l’enzima polinucleotide chinasi che utilizza una molecola di ATP che presenta l’isotopo radioattivo del fosforo (32P) in posizione gamma. La chinasi catalizza la reazione di fosforilazione della sonda all’estremità 5′ libera che risulterà marcata con 32P, liberando una molecola di ADP.
Labeling dell’estremità 3′
Nel caso della marcatura della sonda a livello dell’estremità libera 3’OH, viene sfruttata l’attività dell’enzima DNA nucleotidil-esotransferasi, noto anche come tansferasi terminale. Si tratta di una DNA polimerasi che catalizza l’aggiunta di nucleotidi marcati con isotopi radioattivi all’estremità 3’OH libera della sonda.
Random primer
Il random primer si utilizza qualora non si conosca la sequenza del campione o quando si vogliono produrre numerose copie di corti frammenti di DNA. I random primer sono oligonucleotidi a singolo filamento di DNA composti da 6 nucleotidi la cui sequenza è composta da ogni possibile combinazione di basi. Ciò significa che nella miscela di primer ci sono 46 = 4.096 combinazioni nucleotidiche diverse. Dato che è presente ogni possibile combinazione nucleotidica, i primers riescono a legarsi ad ogni regione della molecola target. Il limite di questa tecnica consiste nell’impiego di oligonucleotidi corti. Essendo composti solo da 6 nucleotidi, in seguito all’ibridazione, i random primer formano pochi legami a ponte idrogeno, per cui l’ibrido è instabile.
Nick translation
Il nick translation è un metodo in grado di marcare il filamento nella sua interezza, non soltanto le estremità 3’OH o 5’P. Si parte da una sequenza di DNA a doppio filamento che viene tagliata da una endonucleasi aspecifica come una DNAsi. L’enzima scinde i legami fosfodiesterici causando rotture a singolo filamento, dette nick. A questo punto interviene la DNA polimerasi I che rimuove i nucleotidi presenti e li sostituisce con deossinucleotidi trifosfati marcati.

Marcatori per le sonde
Traccianti radioattivi
Le sonde possono essere marcate radioattivamente utilizzando gli isotopi radiattivi 32P, 35S, 125I o 3H (tritio). Per ottenere una sonda radiomarcata, si allestisce una reazione di polimerizzazione in vitro in modo che l’isotopo, per esempio 32P, venga incorporato nella sonda. Gli acidi nucleici hanno un’elevato contenuto di fosforo a livello dei legami fosfodiesteri che tengono unite le basi azotate all’interno dello stesso filamento. Alla fine del processo otterremo sonde dove il 32P sostituisce alcuni atomi di fosforo all’interno della catena nucleotidica. Affinché la reazione avvenga, servono i seguenti reagenti:
- molecola di DNA che vogliamo marcare, denaturata
- quattro tipi di deossiribonucleotidi o ribonucleotidi, a seconda che la sonda sia a DNA o RNA, di cui uno marcato con 32P,
- DNA o RNA polimerasi,
- una miscela di oligonucleotidi lunghi 6-9 basi e a sequenza non definita che si appaieranno in maniera casuale sulla molecola di DNA denaturata che servirano da innesco per l’attività della polimerasi.
Il rilevamento delle radiazioni emesse dagli isotopi radioattivi incorporati dalle molecole viene effettuato tramite autoradiografia. Si pone il preparato radiomarcato in contatto con un’emulsione fotografica fotosentibile per un tempo sufficiente affinché la lastra vega impressionata. Quindi, si sfruttano le proprietà ionizzanti delle radiazioni emesse dalle moleocle marcate: tali radiazioni riducono il bromuro di argento presene sull’emulsione fotografica in argento metallico. Si ottiene dunque una lastra fotografica impressionata.
Labeling diretto
Si può marcare la sonda facendole incorporare nucleotidi coniugati a fluorocromi. I fluorocromi più utilizzati sono la fluorsceina che emette nel verde e la rodamina che emette nel rosso. Queste due sostanze sono in grado di emettere fluorescenza anche quando sono presenti in tracce, per cui vengoo utilizzate per rilevare minime quantità di sostanza.
Labeling indiretto
Per labeling indiretto di indente che la sonda che si lega alla molecola target viene riconosciuta da una seconda sonda marcata con fluorocromi o enzimi. Un esempio di labeling indiretto è la marcatura della sonda con il sistema biotina – streptavidina. La biotina è una vitamina (vitamina B8), presente in molti alimenti e che partecipa al metabolismo proteico, alla sintesi degli acidi grassi e del glucosio. Invece, la streptavidina è una proteina extracellulare tetramerica estratta da Streptomyces avidinii caratterizzata da un’alta affinità per la biotina. La tecnica consiste nel far incorporare alla sonda un analogo di un nucleotide contenente biotina mediante random priming o nick translation. La biotina viene riconosciuta dall’avidina accoppiata con fluorocromi o con un enzima.
Analogo al sistema biotina-streptavidina è il metodo di marcatura della sonda con digossigenina. La digossigenina è uno steroide cardiotonico di origine vegetale isolato dalla pianta Digitalis purpurea. La digossigenina incorporata dalla sonda viene riconosciuta da anticorpi marcati e altamente specifici. Il rilevamento del segnare può essere di tipo colorimetrico, fluorescente o luminescente.

Rilevamento per chemioluminescenza
Qualora la molecola specifica per la biotina o digossigenina venga associata a un enzima, bisogna aggiungere il suo substrato e coenzima in soluzione. Al fine di rilevare l’ibrido sonda-molecola target, si utilizza un substrato cromogenico che diventi fluorescente o luminescente in seguito alla reazione catalizzata dall’enzima. In questi casi, spesso la sonda è complessata con la perossidasi di rafano (Horseradish peroxidase) e il substrato selezionato è il luminolo. Si tratta di una ossidoreduttasi che, in presenza di acqua ossigenata, riduce il luminolo in un composto in grado di sviluppare chemioluminescenza.

La tecnica dell’ibridazione
La sonda deve essere messa in contatto con il gene target per poter sfruttare la tendenza degli acidi nucleici di appaiarsi con le sequenze complementari. Questo processo di riconoscimento e appaiamento (ibridazione) può avvenire solo in seguito alla denaturazione delle molecole a doppio filamento. Affinché le due catene della molecola di DNA si destabilizzino e si separino, devono verificarsi le seguenti condizioni:
- Temperature alte (T > 90°C)
- pH alcalino estremo (pH >13)
- Bassa forza ionica [Na+]
- Presenza in soluzione di sostanze denaturanti che rompono i legami a idrogeno (urea, formammide)
Dopo aver denaturato le molecole di DNA è necessario ridurre gradualmente la temperatura. Dato che i moti di agitazione termica influiscono su processo di ibridazione, le molecole devono restare pochi gradi al di sotto della temperatura di denaturazione. Se dopo la denaturazione si porta la soluzione bruscamente a basse temperature non si ha ibridazione, ma stabilizzazione della struttura secondaria delle singole catene. L’ibridazione può avvenire tra due filamenti di RNA o tra DNA e RNA. L’ibrido RNA/RNA e RNA/DNA è più stabile ispetto all’ibrido DNA/DNA. Il filamento utilizzato per l’ibridizzazione di un DNA bersaglio può essere un innesco (primer), necessario per dare via all’amplificazione, o una sonda.
Tecniche che sfruttano il labeling degli acidi nucleici
Saggi di ibridazione standard
Dot blot
Per Dot blot si intende una tecnica biochimica che evidenzia la presenza di un gene all’interno di uno specifico organismo sfruttando l’ibridazione DNA/DNA. Questa tecnica rappresenta una semplificazione del Western blot che ne riduce i tempi e i costi. Basta pensare che le proteine non vengono prima separate per elettroforesi su gel: si applica il campione direttamente sulla membrana in un unico punto. Il dot blot prevede la fissazione delle cellule del campione su un filtro di nylon o nitrocellulosa, che viene posto su della carta assorbente. Dopo aver lisato le cellule, si fissa il DNA sul supporto e si induce la formazione di dimeri di timina tramite esposizione ai raggi ultravioletti. Infine, si denatura tramite l’aggiunta di soda caustica e si inseriscono le sonde a DNA marcate con un fluorocromo. Le sonde si legheranno alla loro sequenza complementare e dopo autoradiografia evidenzieranno quali cellule possiedono il gene di interesse.
Solitamente si usa il Dot blot nella diagnostica microbiologica perché consente di effettuare rapidi screening alla ricerca di patogeni, come Chlamydia trachomatis. La tecnica dot blot può essere anche utilizzata per confermare la presenza di Listeria monocytogenes nel cibo.

Southern blot
Il Southern blot è una tecnica utilizzata in biologia molecolare per rilevare la presenza di specifiche sequenze di DNA all’interno di un campione. Brevemente, il campione digerito dagli enzimi di restrizione viene sottoposto ad elettroforesi su gel. Successivamente, si immerge il gel in una soluzione alcalina al fine di denaturare il DNA. Poi, si copre il gel con una membrana di nylon o nitrocellulosa (carica positivamente), della carta assorbente e un peso. La soluzione si muoverà per capillarità verso l’alto depositando il DNA (carico negativamente) sulla membrana, con la quale formerà dei legami elettrostatici. A questo punto, si saturano le cariche aggiungedo del DNA eterologo per saturare le cariche e si incuba la membrana con sonde radiomarcate. La rilevazione viene eseguita mediante autoradiografia. Si può ricorrere al Southern blot per i seguenti scopi:
- studiare la struttura e l’organizzazione di un gene,
- charire le mappe di restrizione,
- identificare i siti metilati di determinati geni,
- controllare eventuali organismi geneticamente modificarti (OGM),
- diagnosticare malattie genetiche (fibrosi cistica, distrofia muscolare…),
- individuazione di agenti patogeni nelle infezioni,
- analisi filogenetiche,
- analisi forensi.

Northern blot
Il Northern blot è utilizzato per identificare le molecole di RNA purificate dal campione. Si tratta di una tecnica analoga al Southern blot, con la differenza che l’RNA deve essere prima denaturato al fine di eventuali strutture secondarie. Inoltre, la corsa elettroforetica deve avvenire in presenza di agenti denaturanti, come formaldeide e formammide. Il Northern blot si usa principalmente per studiare l’espressione genica e per determinare la dimensione e la quantità di determinati RNA.
FISH
L’ibridazione fluorescente in situ o FISH (Fluorescent In Situ Hybridization) è una tecnica di ibridazione su tessuti o cromosomi. Si tratta di una tecnica utilizzata in citogenetica molecolare che fa uso di sonde a DNA fluorescenti. Legandosi per complementarietà a specifiche sequenze, la sonda consente di identificare la posizione relativa di tali sequenze sui cromosomi. Per individuare il sito di legame della sonda sul cromosoma si utilizza il microscopio a fluorescenza.
Si possono utilizzare molteplici sonde, ognuna marcata con un fluorocromo differente, che corrispondano a frammenti specifici di un singolo cromosoma. Questa tecnica viene chiamata FISH multicolore. Una variante del FISH multicolore consiste nell’utilizzo di sonde marcate con fluorocromi diversi, ognuna specifica per un singolo cromosoma. Così si ottiene una mappa in cui ogni cromosoma è fluorescente ed ha un colore diverso. La FISH può essere applicata per diversi scopi, in particolare per rilevare eventuali anomalie numeriche e strutturali dei cromosomi associate a patologie.

Saggi di ibridazione inversi
Per saggi di ibridazione inversi si intendono quelle tecniche che utilizzano una sonda genetica non marcata e legata a un supporto solido. Bensì, si marca la sequenza target che si trova in soluzione.
Microarray
I microarray o gene chip sono sonde fissate su un supporto di vetro, silicio o plastica formando una matrice (array). Ciò che si marca in questa tecnica sono le molecole di DNA che vogliamo esaminare, generalmente tramite l’impiego di un fluorocromo. Questa tecnica permette di esaminare simultaneamente numerosi geni all’interno di un campione di DNA che può rappresentare l’intero genoma o trascrittoma dell’individuo. Per studiare l’espressione genica di un individuo basta estrarre i suoi RNA e retrotrascriverli in cDNA per mezzo di una trascrittasi inversa. Una volte che è avvenuta l’ibridazione campione marcato-sonda, si procede con la visualizzazione e la quantificazione del segnale fluorescente legato a ciascuna sonda. L’intensità del segnale fluorescente è proporzionale alla quantità di sequenza complementare che si è legata alla sonda. Il microarray si può utilizzare per confrontare il profilo di espressione genica del paziente con un controllo proveniente da un individuo sano.

Applicazioni delle tecniche di labeling
Il labeling è una tecnica di fondamentale importanza per la ricerca e per le applicazioni biomediche e biotecnologiche. In particolare, la marcatura e l’ibridazione degli aicdi nucleici con le sonde sono indispensabili per l’identificazione di geni all’interno di librerie genomiche o di cDNA. In medicina, le tecniche di labeling permettono l’individuazione di microrganismi o la diagnosi di determinate patologie.
Le tecniche di labeling sono fondamentali anche in ambito forense. Per fare un esempio, si può confrontare il materiale genetico presente sulla scena del crimine con quello dell’indiziato. Si impiegano endonucleasi per frammentare i campioni in RFLP (polimorfismo di lunghezza dei frammenti di restrizione). Tali frammenti si separano tramite corsa elettroforetica su gel, dopodiché un’apposita sonda radioattiva si lega ai RFLP e la rilevazione si effettua per autoradiografia. Se le sequenze delle bande ricavate dal campione e dall’indiziato coincidono, si calcola la probabilità con la quale tale corrispondenza sia solo accidentale.
Fonti
- https://www.treccani.it/enciclopedia/sonde-molecolari_%28Enciclopedia-Italiana%29/
- https://moodle2.units.it/pluginfile.php/175833/mod_resource/content/1/Lezione%2010.pdf
- https://it.wikipedia.org/wiki/Southern_blot
- https://it.wikipedia.org/wiki/Dot_blot
- https://it.wikipedia.org/wiki/Microarray_di_DNA
Crediti immagini
- Immagine in evidenza: https://en-med.tau.ac.il/safety_biology_ethidium_bromide-2020
- Figura 1: https://slideplayer.it/slide/13801839/
- Figura 2: https://www.aatbio.com/products/readilink-biotin-nick-translation-dsdna-labeling-kit
- Figura 3: https://moodle2.units.it/pluginfile.php/175833/mod_resource/content/1/Lezione%2010.pdf
- Figura 4: https://www.researchgate.net/publication/345718018_Existing_Biosensing_Platforms_for_the_Nucleotide_Markers_Detection
- Figura 5: http://novateinbio.com/techinfo/protocols/Dot%20Blot%20Protocol.pdf
- Figura 6: https://www.thesciencenotes.com/southern-blotting-principle-procedure-and-applications/
- Figura 7: http://feedforbiotech.blogspot.com/2011/03/chromosome-painting.html
- Figura 7: https://sites.google.com/site/suneelszoology/instruments-techniques/chromosome-painting
- Figura 8: https://www.diagnosiprenatale.com/diagnosi-prenatale/cariotipo-molecolare-tecnica-array-cgh.aspx