Sequenziamento Sanger
Il sequenziamento del DNA è diventato una procedura abituale all’interno dei laboratori d’analisi in maniera particolare durante la pandemia di Sars-Cov 2. Possiamo raggruppare le tecniche di sequenziamento in tre categorie:
- Sequenziamento di prima generazione
- Next generation sequencing o sequenziamento di seconda generazione (NGS)
- Sequenziamento di terza generazione o Third generation sequencing
Nel seguente articolo parleremo della prima tecnologia di sequenziamento ideata dallo scienziato Frederick Sanger grazie alla quale conseguì il premio Nobel per la Chimica nel 1980. (Figura 1)

Caratteristiche generali
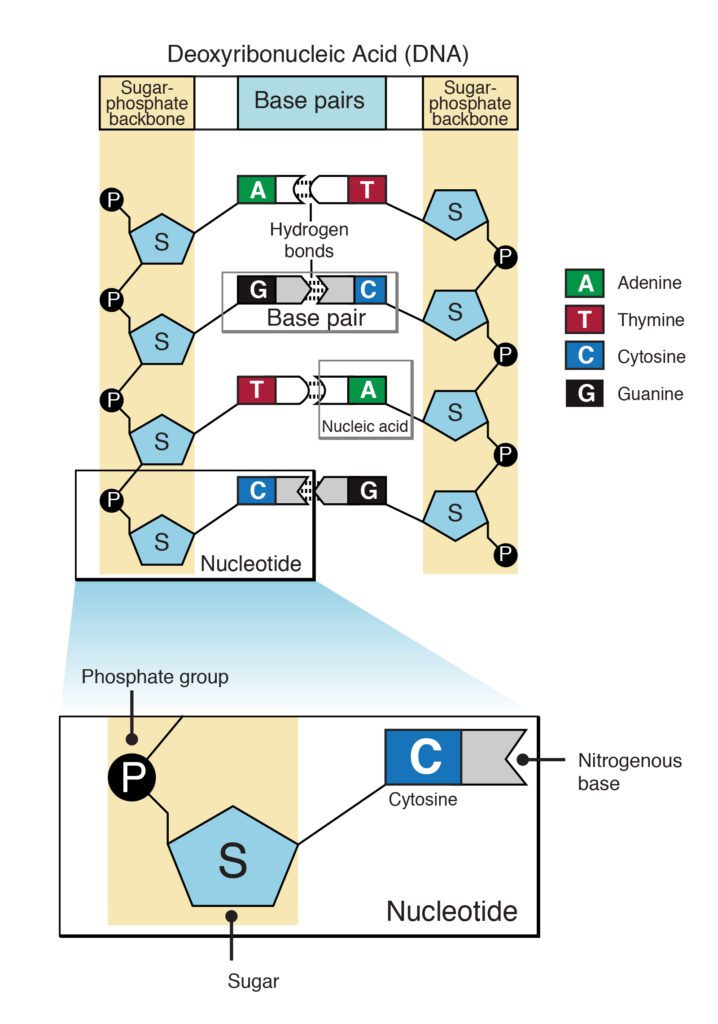
Per poter comprendere al meglio il funzionamento della tecnica di Sanger è necessario ripassare in breve qualche concetto di biochimica sulla struttura del DNA o acido deossiribonucleico. Il DNA è un polimero costituito da più monomeri definiti nucleotidi. Ogni nucleotide è costituito dalle componenti indicate nella seguente tabella. (Tabella 1)
| Zucchero pentoso | Il deossiribosio è lo zucchero pentoso presente all’interno del DNA |
| Base azotata | Ogni nucleotide è distinguibile dall’altro grazie alla presenza di una base azotata, nel DNA ci sono quattro tipi di basi: adenina (A), timina (T), guanina (G) e citosina (C) |
| Gruppo fosfato | Fornisce al DNA proprietà chimiche acide a pH fisiologico. |
I nucleotidi vanno a costituire lo scheletro carbonioso del DNA legandosi al nucleotide adiacente attraverso un legame fosfodiesterico mentre le basi si appaiano con il filamento antiparallelo attraverso i legami idrogeno che avvengono tra le basi azotate. Tra adenina e timina si formano due legami idrogeno mentre tra guanina e citosina se ne formano tre di legami di idrogeno. (Figura 2)
Funzionamento del Sequenziamento Sanger
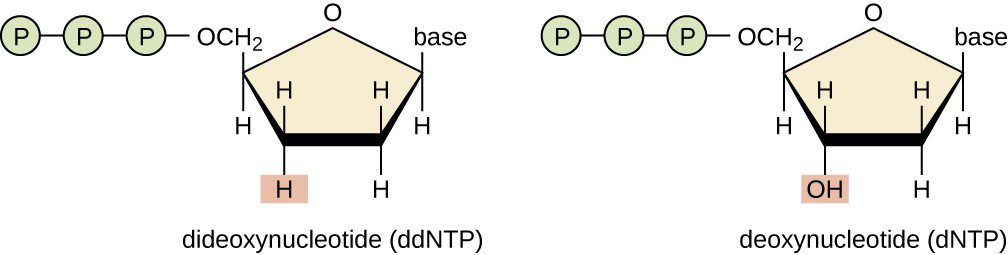
Il metodo di Sanger o metodo della terminazione della catena consiste nell’utilizzo di dideossiribonucleotidi ( struttura a sinistra, Figura 3); quest’ultimi sono privi del gruppo ossidrilico in posizione 3′. Quando un dideossiribonucleotide viene aggiunto alla catena da una DNA polimerasi ad alta processività questo comporta la cessazione dell’allungamento della catena perché non può avvenire la formazione del legame fosfodiesterico.
Per il sequenziamento di un frammento di nostro interesse, opportunamente amplificato, prepariamo 4 reazioni di PCR, ognuna delle quali conterrà le seguenti componenti:
- Frammento di DNA da sequenziare denaturato;
- Primer, una sequenza oligonucleotidica, complementare alla regione fiancheggiante al frammento di DNA da sequenziare e marcato radioattivamente;
- DNA polimerasi ad alta processività;
- 4 dNTP, deossiribonucleotidi trifosfati;
- Un ddNTP dideossiribonucleotide trifosfato.
Procedimento
Suddividiamo la nostra miscela in quattro frazioni per ogni base nucleotidica (A, T, G, C) alla quale aggiungiamo il terminatore di catena corrispondente (ddATP, ddTTP, ddGTP, ddCTP). L’aggiunta di questi terminatori di catena è puramente causale quindi si formeranno frammenti di lunghezza diversa che avranno tutti come sequenza iniziale quella del premier marcato e termineranno con il dideossiribonucleotide trifosfato.
Dopo l’incubazione le quattro frazioni vengono denaturate al calore, per separare le catene nucleotidiche appaiate, e sottoposte a corsa elettroforetica in un unico gel di poliacrilammide. Le catene neosintetizzate migrano nel gel in base alla loro lunghezza, quelle più piccole tenderanno a spostarsi più velocemente rispetto a quelle più lunghe; inoltre sarà possibile individuarle facilmente perché marcate radioattivamente. (Figura 4)
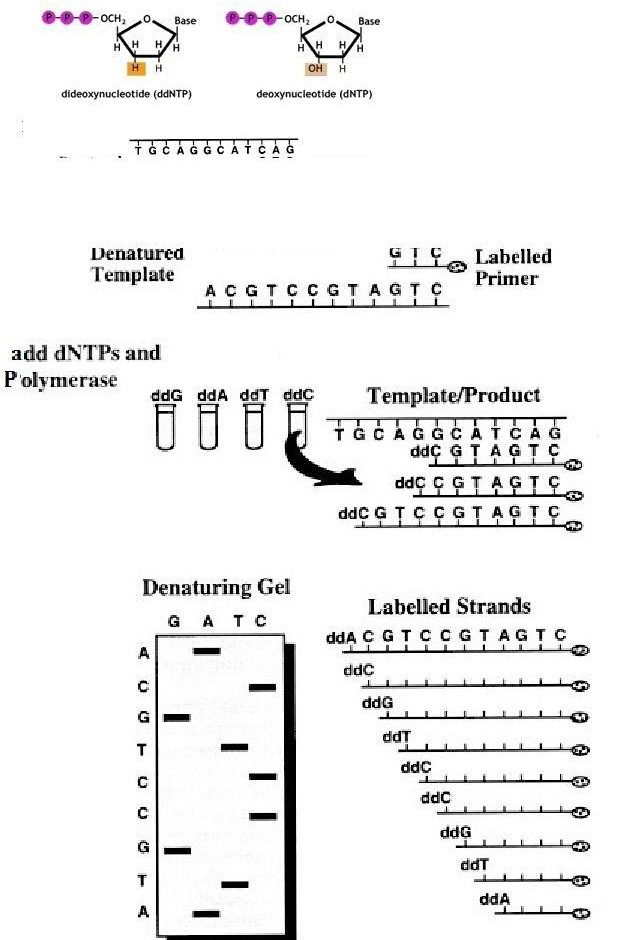
Questa condizione si verificherà anche nelle altre quattro corsie del gel e dalla successione di tutte le bande presenti nelle quattro corsie del gel sarà possibile risalire alla sequenza nucleotidica, che viene ricostruita dall’estremità 5′,del frammento di DNA usato come stampo.
Limitazioni del sequenziamento Sanger
Oramai questa tecnologia è stata superata dal metodo di Sanger automatizzato. Questo metodo richiede molto lavoro e tempo, infatti sono necessari l’impiego di grandi quantità gel di poliacrilammide , lo sviluppo di lastre da esporre ai raggi X; in più è richiesto l’impiego di radioisotopi e la determinazione della sequenza viene effettuata manualmente. Attraverso l’uso di questa tecnica è possibile sequenziare 200 – 300 paia di basi in 2 – 3 giorni.
Metodo Sanger automatizzato
Il metodo di Sanger nel corso del tempo ha subito dei miglioramenti, infatti oggi si parla di metodo Sanger automatizzato. Questa tecnica non prevede l’utilizzo di radioisotopi ma l’utilizzo di 4 fluorocromi differenti legati ai terminatori di catena così da non dover ricorrere all’uso di quattro reazioni separate. La corsa elettroforetica avviene all’interno di un gel elettroforetico di forma cilindrica, sempre in base alla dimensione dei frammenti neosintetizzati.

La lettura non avviene manualmente ma si avvale dell’utilizzo di un laser che eccita i fluorocromi, le emissioni di luce vengono poi rilevate da un detector. Dalle informazioni ottenute e registrate otterremo un cromatogramma dove saranno presenti differenti picchi di colore in base all’intensità dell’emissione di luce. Attraverso questo metodo è possibile sequenziare un frammento di DNA che contiene dalle 700 alle 1000 paia di basi in poche ore. (Figura 5 )
Limitazioni/ Limiti della tecnica
Nonostante il metodo di Sanger automatizzato permetta di sequenziare un elevato numero di basi in poche ore rimane una tecnica di sequenziamento costosa se paragonata a quelle della Next generation sequencing.
Fonti
- “Principi di Genetica” – V edizione di D.P. Snustad e M. J Simmons. Edises. ISBN 978 88 7959 839 2
Fonti immagini
- https://it.wikipedia.org
- https://www.genome.gov
- https://www.genomeup.com
- https://www.khanacademy.org
- https://gioaboffa.wordpress.com
Ismael Sanchez Polanco