Mutazioni del DNA: caratteristiche generali
Il DNA è un acido nucleico che contiene l’informazione genetica per la biosintesi di RNA e proteine. Si duplica, viene trascritto e subisce mutazioni, ovvero modificazioni covalenti a livello dei nucleotidi causati da:
- errori durante la duplicazione
- danni chimico-fisici al DNA
- spostamento di trasposoni nel genoma
Le mutazioni in una frequenza alta sono un fattore negativo per le cellule. Esse possiedono, infatti, dei meccanismi di riparazione del DNA per evitare un accumulo eccessivo di mutazioni che concorrono alla formazione di cellule tumorali.
Tipologie di mutazioni a carico del DNA e rispettivi metodi di riparazione
Vediamo adesso le classi mutazionali esistenti e come vengono riparate più nel dettaglio.
1) Mismatch: errori durante la replicazione e loro correzione
Durante la sintesi del DNA possono verificarsi degli appaiamenti non complementari tra le basi azotate dando luogo ad un mismatch. Esso è dovuto principalmente alla tautomeria, uno spostamento di elettroni che converte una base azotata in un’altra.
Un esempio è la tautomeria cheto-enolica della timina che passa dalla forma chetonica a quella enolica a seguito di spostamenti elettronici nelle posizioni C3 e C4. In questa nuova conformazione la timina sarà complementare alla guanina.
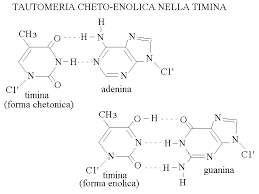
L’Adenina, invece, è interessata da una tautomeria ammino-imminica che coinvolge le posizioni 1 e 6 risultando così complementare alla Citosina.
Nel momento in cui una base azotata assume una diversa conformazione e viene inserita nella doppia elica si ha un mismatch, che può essere riparato tramite due principali vie:
- Proof-reading: attività esonucleasica insita alla DNA polimerasi III (enzima deputato alla duplicazione del DNA) che riconosce il mismatch, blocca la sintesi e torna indietro nel filamento in direzione 3’-5’ digerendo una piccola sequenza di basi comprendente l’errore. Si forma una giunzione innesco-stampo con estremità 3’OH libera rendendo possibile la sintesi con l’innesco del dDNTP giusto. Si riduce la probabilità di errore di 10-7 ;
- Proof-reading post sintetico/ Mismatch Repair (MMR): si attiva nel caso in cui all’attività esonucleasica della DNA polimerasi sfugga l’errore. Il sistema MMR è attuato da una serie di proteine MUT che agiscono da onco-soppressori a livello del filamento neo-sintetizzato, prima che esso sia sottoposto a metilazione (meccanismo di regolazione della trascrizione). La presenza del mismatch induce una perdita di parallelismo della doppia elica che viene riconosciuto dalle proteine MUT, in particolare dal tipo MutS, che si lega al nuovo filamento in prossimità dell’errore. L’altra proteina MutL si inserirà poi nel filamento stampo in corrispondenza di un sito metilato vicino al mismatch e di conseguenza a MutS. A questo punto interviene MutH, un’endonucleasi che forma un nick sul filamento neo-sintetizzato e a seguire una esonucleasi che darà luogo ad un gap risanato poi da una DNA-polimerasi ed una ligasi, ricostruendo così la sequenza corretta.
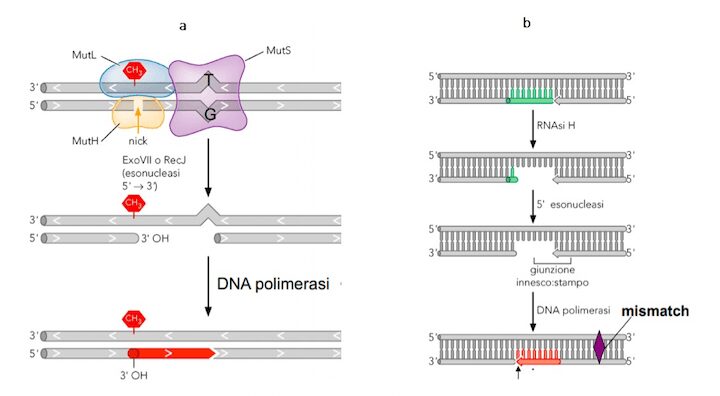
2) Modificazioni chimiche spontanee e loro riparazione
Per mutazioni chimiche spontanee delle basi azotate si intendono quei processi di deaminazione, alchilazione, ossidazione ed idrolisi del gruppo amminico che trasformano chimicamente la complementarietà di una base azotata. Il mismatch che si forma sarà riparato tramite metodi diretti o indiretti.
Come ci dice il nome, la deaminazione consiste nella perdita del gruppo amminico NH2 della Citosina che si trasforma in Uracile, a sua volta complementare all’adenina. L’Adenina deaminata, invece, si trasformerà in ipoxantina che è complementare alla citosina e la guanina che subisce deaminazione diventa xantina, a sua volta affine alla timina.
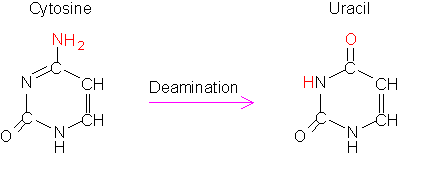
L’ossidazione interessa principalmente la guanina a livello del C in posizione 8 diventando 8-oxoguanina, complementare alla timina.
L’idrolisi del gruppo amminico della citosina produce deossi-uracile, che forma legami a idrogeno con l’Adenina.
Tutte queste modifiche chimiche spontanee creano dei mismatch che vengono riparati in modo diretto tramite il meccanismo BER/ riparazione per escissione di base, che consiste nell’eliminazione della sola base azotata, preservando lo scheletro zucchero-fosfato del nucleotoide. Ciò è possibile grazie all’enzima glicolasi,che riconosce la mutazione rompendo il legame glicosidico tra base e zucchero, creando un sito abasico in cui interviene l’endonucleasi A1 per la formazione di un nick. Esso diventa sito di attacco per una esonucleasi che dà luogo ad un gap risanato poi da DNA-polimerasi e ligasi.
Esistono altre modificazioni chimiche riparate in modo diretto, ovvero tramite formazione spontanea del sito abasico ed è questo il caso di mismatch a seguito di:
- alchilazioni delle basi: la guanina metilata, ad esempio, si trasforma in O-metil-guanina, che sarà complementare alla timina. A seguito di tale trasformazione entra in gioco l’enzima metil-transferasi, contenente una cisteina attiva che strappa il metile alla base modificata ottenendo la guanina originale;
- idrolisi spontanea del legame glicosidico: la rottura di tale legame produce direttamente un sito abasico riparato dalla sequenza vista precedentemente (endonucleasi-esonucleasi- Dna polimerasi e ligasi).
3) Modificazioni chimiche indotte e loro riparazione
Un esempio di modificazione chimica indotta covalente è la formazione di dimeri di timina a seguito dell’esposizione a raggi UV, che può essere riparato tramite i seguenti meccanismi:
- fotoliasi luce dipendente: funziona in presenza della luce stessa, grazie all’energia delle radiazioni elettromagnetiche sfruttata per rompere l’anello ciclo-butanico tra le due timine (dimero).
- sistema UVR: facente parte del meccanismo NER (riparazione per escissione di nucleotidi) che prevede l’interazione di proteine del complesso UVrAB. Esso si attiva scindendosi in UVrB che si posiziona nel dimero aprendo la doppia elica e in UVrA che crea una distorsione del filamento rompendo i legami a idrogeno. SI attiva anche UVrC, una endonucleasi a due domini, che crea due nick trasformati in gap dalla elicasi, poi risanati da DNA-polimerasi e ligasi.

- Sintesi trans-lesione: se i precedenti meccanismi falliscono, il dimero viene mantenuto e ciò causa un impedimento della replicazione del filamento di DNA a seguito del grande ingombro sterico, inducendo la progressiva apoptosi della cellula. Infatti, esiste la DNA-polimerasi LTS: enzima dotato di un sito attivo più grande, che riesce ad oltrepassare il dimero a differenza della DNA-polimerasi III di dimensioni ridotte. L’enzima LTS permette di salvare la cellula dall’apoptosi ma, avendo uno scarso proof-reading, spesso dà luogo a mutazioni del DNA puntiformi, in cui ad una delle due timine viene accoppiata una guanina.
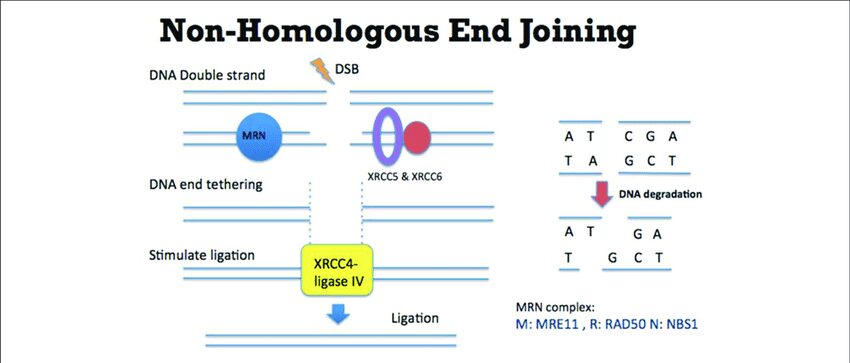
L’esposizione a radiazioni o mutageni chimici può indurre anche un altro danno al DNA causativo di mutazioni: il double strand break (DSB).
Esso consiste in una rottura della doppia elica, con esposizione di due estremità libere legate dalle elicasi KU con lo scopo di cercare zone di micro-omologia e creare due giunzioni innesco-stampo usate da una DNA-polimerasi per ristabilire la continuità. Ciò comporta, in ogni caso, delezione di alcune basi, con spostamento della griglia di lettura (mutazione di frame-shift) che, se fondamentali, inducono un grave danno fenotipico. Questo meccanismo nei mammiferi si chiama NHEJ/ Non Homologus Ended Joining.
A questo proposito, esiste anche un altro meccanismo di riparazione simile, definito HR/ Homologus Recombination riguardante le cellule diploidi. Si basa sul ricopiare le informazioni dall’allele normale su quello in cui è presente un DSB, tramite un fenomeno di ibridazione e ricombinazione.
4) Mutazioni del DNA dovute allo spostamento di elementi trasponibili
I trasposoni sono sequenze di DNA in grado di spostarsi da un punto all’altro del genoma modificando il corredo genetico di un essere vivente. Infatti, gli effetti della trasposizione sono traslocazione o interruzione di cluster genici ,con conseguenti alterazioni fenotipiche dovute ad acquisizione di nuove funzioni o morte della cellula stessa.
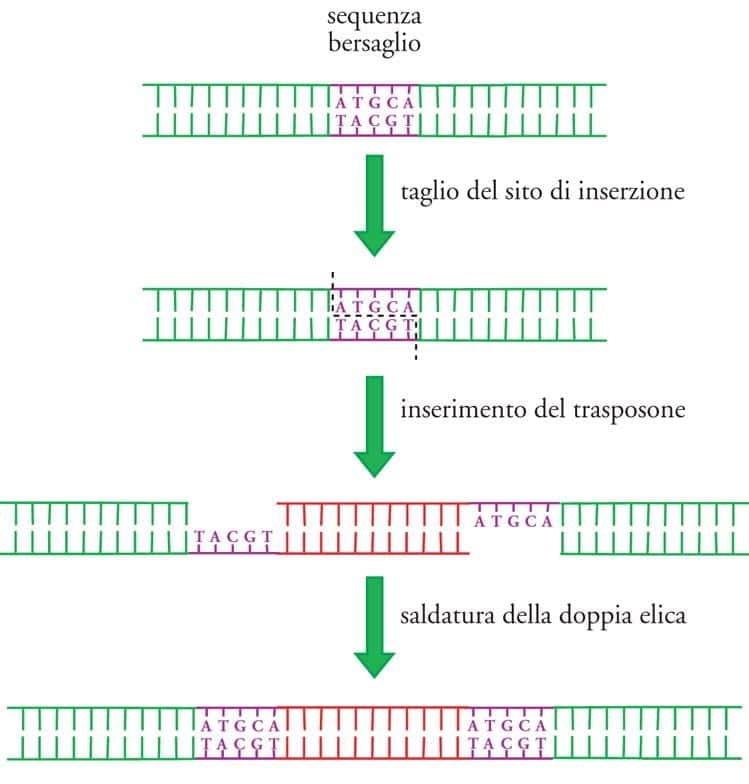
Conseguenze biologiche
Abbiamo visto le mutazioni del DNA più frequenti ed i rispettivi meccanismi di riparazione. Queste sono molto efficienti e assicurano un basso tasso mutazionale negli eucarioti; tuttavia, può comunque succedere che alcuni mismatch non vengano riparati e, di conseguenza, fissati in mutazioni permanenti che si riflettono anche nel prodotto proteico visibile nel fenotipo. Se queste modificazioni interessano geni non fondamentali, le conseguenze sono trascurabili ma, se invece colpiscono onco-soppressori ed onco-geni, concorrono alla formazione di tumori. Una mutazione iper-attivante in eterozigosi su un onco-gene è di fatti sufficiente all’innesco della tumorigenesi.
Allo stesso tempo, però, sono state proprio le mutazioni genetiche ad indurre l’evoluzione della vita sulla Terra.
Test diagnostici
Attualmente, le mutazioni del DNA possono essere facilmente identificate mediante test genetici. E’ sufficiente un prelievo di sangue venoso o di altri fluidi biologici, da cui è possibile estrarre il DNA. Esso sarà amplificato tramite Real-time PCR e analizzato per mezzo di vari meccanismi di screening (es. next generation sequencing, analisi con enzimi di restrizione…)
Un test genetico diventa molto importante in quei casi di familiarità per tumori o malattie genetiche in cui ricorre nell’albero genealogico una determinata mutazione.
Fonti
- https://pubmed.ncbi.nlm.nih.gov/29688526/
- https://pubmed.ncbi.nlm.nih.gov/1369117/
- https://pubmed.ncbi.nlm.nih.gov/2016084/
- https://pubmed.ncbi.nlm.nih.gov/8875255/
- https://www.sciencedirect.com/science/article/abs/pii/S1383571822000900
- https://pubmed.ncbi.nlm.nih.gov/8393197/
Crediti immagini
- immagine in evidenza: https://www.issalute.it/index.php/la-salute-dalla-a-alla-z-menu/m/
- Figura 1: http://biochimica.bio.uniroma1.it/didattica/organica/glicidi.htm
- Figura 2: https://www.chimica-online.it/biologia/riparazione-del-dna.htm
- Figura 3: https://www.chimica-online.it/composti-organici/citosina.htm
- Figura 4: https://www.microbiologiaitalia.it/didattica/meccanismi-di-riparazione-dei-danni-al-dna/
- Figura 5: https://www.researchgate.net/figure/Mechanism-of-Non-Homologous-End-Joining-NHEJ-Left-DSB-ends-are-tethered-by-MRN_fig1_327044002
- Figura 6: https://www.biopills.net/trasposoni/