La trascrizione indotta da Tetraciclina, detta Tet-On Tet-Off, è un metodo per controllare l’espressione genica in cellule eucariotiche, sia in ambito di ricerca, sia per fare terapia genica. Questo sistema si basa sull’attività dell’operone procariotico responsabile della resistenza alla tetraciclina.
Cenni storici del sistema Tet-On/Tet-Off e la sua funzione in Escherichia Coli
Il sistema Tet-On/Tet-Off venne messo a punto da Manfred Gossen e Hermann Bujard nel 1992 presso l’Università of Heidelberg (Germania). I due ricercatori pubblicarono quell’anno un articolo sull’utilizzo in cellule di mammifero di un meccanismo naturalmente presente in Escherichia Coli come strumento di difesa verso un antibiotico: la tetraciclina. Questo farmaco è infatti capace di legare la subunità 30S dei ribosomi batterici bloccandone la traduzione proteica. Tuttavia, grazie al meccanismo di resistenza si instaura nella cellula batterica un efflusso dell’antibiotico verso l’esterno della cellula, permettendo così di eludere l’attività del farmaco.
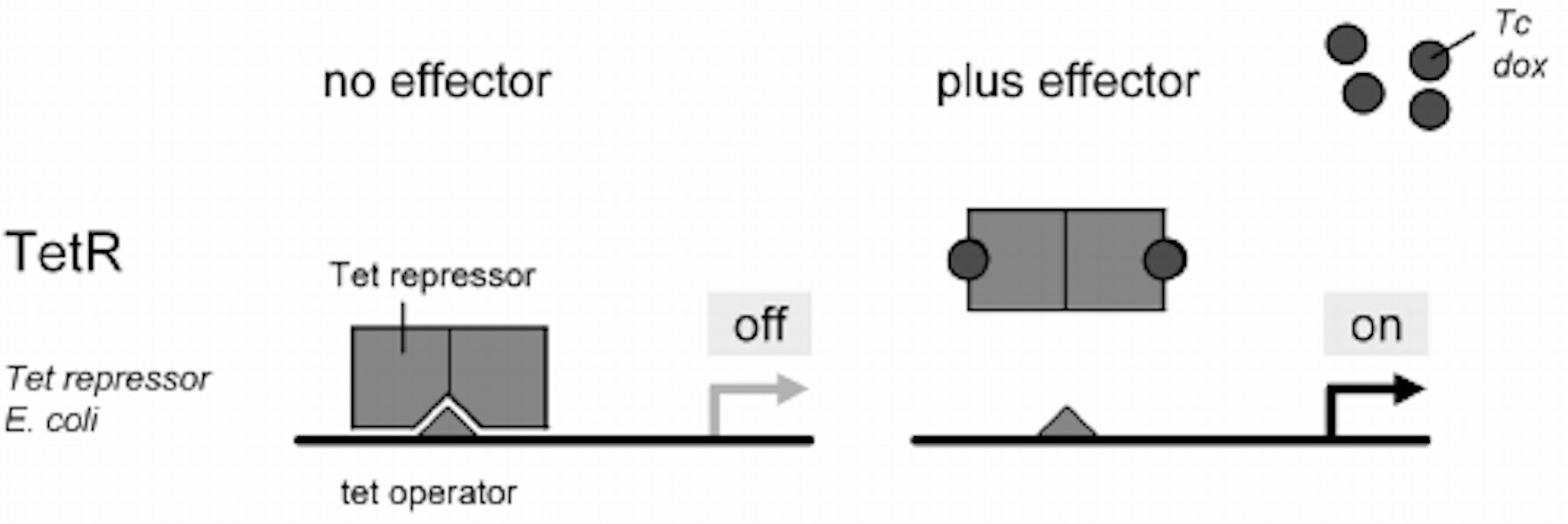
Questo meccanismo si trova nel trasposone Tn10 di E.Coli negativamente controllato da una proteina detta tetR (repressore della tetraciclina-resistenza). In base alla struttura tridimensionale è capace di legare il promotore a monte dei geni per la resistenza (in un sito detto tetO, tet-Operator) inibendo la trascrizione genica. Tuttavia, tale struttura è influenzabile dalla presenza o meno della tetraciclina (o l’analogo doxiciclina). Solo in assenza di tetraciclina tetR ha una conformazione tale da legare tetO (spegnendo la trascrizione) (figura 1). Questo è necessario perché per la cellula batterica sarebbe uno svantaggio sintetizzare le proteine per la resistenza al farmaco in sua assenza. Viceversa, in presenza di tetraciclina, tetR assume una struttura che non le permette più di legare tetO attivando così la trascrizione genica.
Sistema Tet-off
Il sistema Tet-off è stato il primo a essere messo a punto e anche il più noto, perciò iniziamo la nostra descrizione da questo. Per descrivere al meglio l’intero meccanismo distinguiamo tre momenti fondamentali nel protocollo di lavoro di Tet-off, che andremo poi a descrivere in sotto-paragrafi dedicati.
- Trasfezione delle cellule target eucariotica con il vettore plasmidico pTet-off. Selezione delle cellule trasfettate mediante il gene di resistenza per G418 (geneticina, ovvero un antibiotico) contenuto nel vettore stesso.
- Trasfezione delle cellule pTet-off positive con il plasmide pTRE-GOI e selezione con un altro gene di resistenza a antibiotici.
- Utilizzo di tetraciclina (o dell’analogo doxiciclina) per variare i livelli di espressione del gene di interesse.
1. Trasfezione di pTet-off
Nel sistema Tet-off la proteina procariotica tetR si ritrova nel contesto di in una proteina di fusione di 37kDa, frutto dell’unione di tetR con il dominio attivatore della proteina 16 di Herpes Simplex virus, detta VP16. La proteina che si genera, detta tTA, acquista così la funzione di attivatore trascrizionale inducibile con tetraciclina (o l’analogo meno tossico doxiciclina). Questa informazione viene portata dal vettore plasmidico pTet-Off (figura 2), dove tTA (tetR + VP16) è sotto un promotore costitutivamente attivo come quello di Citomegalovirus (PCMV). In più, a seguito del dominio VP16 viene aggiunta una sequenza di poli-adenine che aumenta la stabilità dell’mRNA.
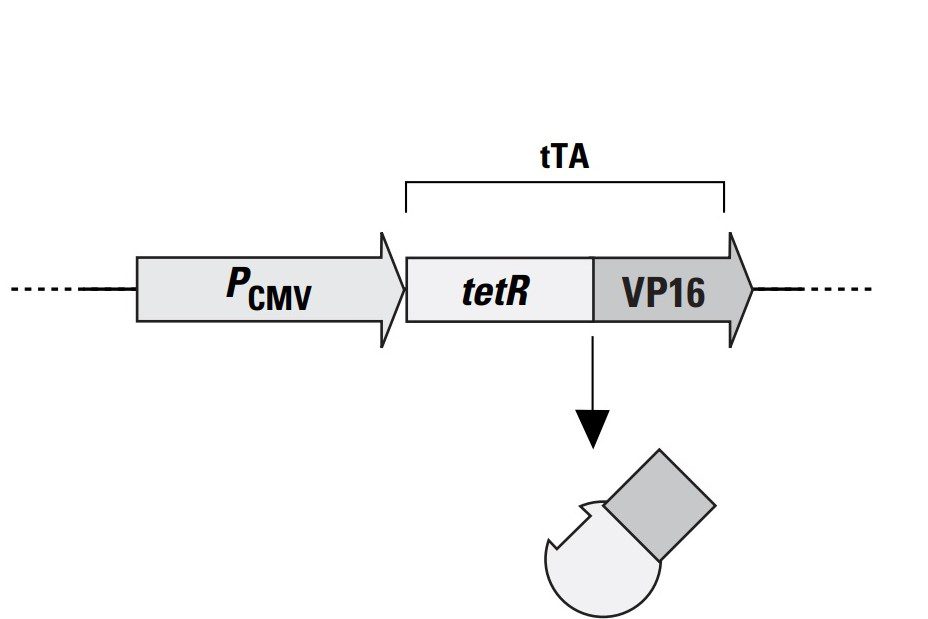
Da notare che il plasmide pTet-Off si costruisce e amplifica in E.Coli prima di essere trasfettato nelle cellule target. Infatti questo vettore, oltre agli elementi sopracitati che servono alla cellula eucariotica, contiene anche un’origine di replicazione batterica e un gene di resistenza all’ampicillina. Solo dopo aver selezionato le cellule batteriche contenenti il vettore e averlo amplificato in sufficienti copie si prosegue con la trasfezione in cellule target.
2. Trasfezione di pTRE-GOI
Secondo elemento fondamentale per la buona riuscita del sistema tet-off è la trasfezione del plasmide pTRE-GOI (Gene Of Interest) (figura 3). Qui si trova il nostro gene di interesse che viene posto sotto il controllo di un elemento regolatore della trascrizione detto TRE (tetracycline-response element). Questa sequenza è costituita da 7 ripetizioni del dominio tetO (ricordiamo può essere legato da tetR e quindi da tTA) e collocata immediatamente a monte del promotore, generalmente del tipo P-CMV. Questo viene privato di alcune sequenze enhancer così che abbia una trascrizione basale quasi assente, a meno che la regione TRE non si attivi.

[Fonte: https://www.takarabio.com/documents/User%20Manual/PT3001/PT3001-1_100912.pdf]
3. Aggiunta di Tetraciclina (o Doxiciclina) per regolare i livelli di espressione genica
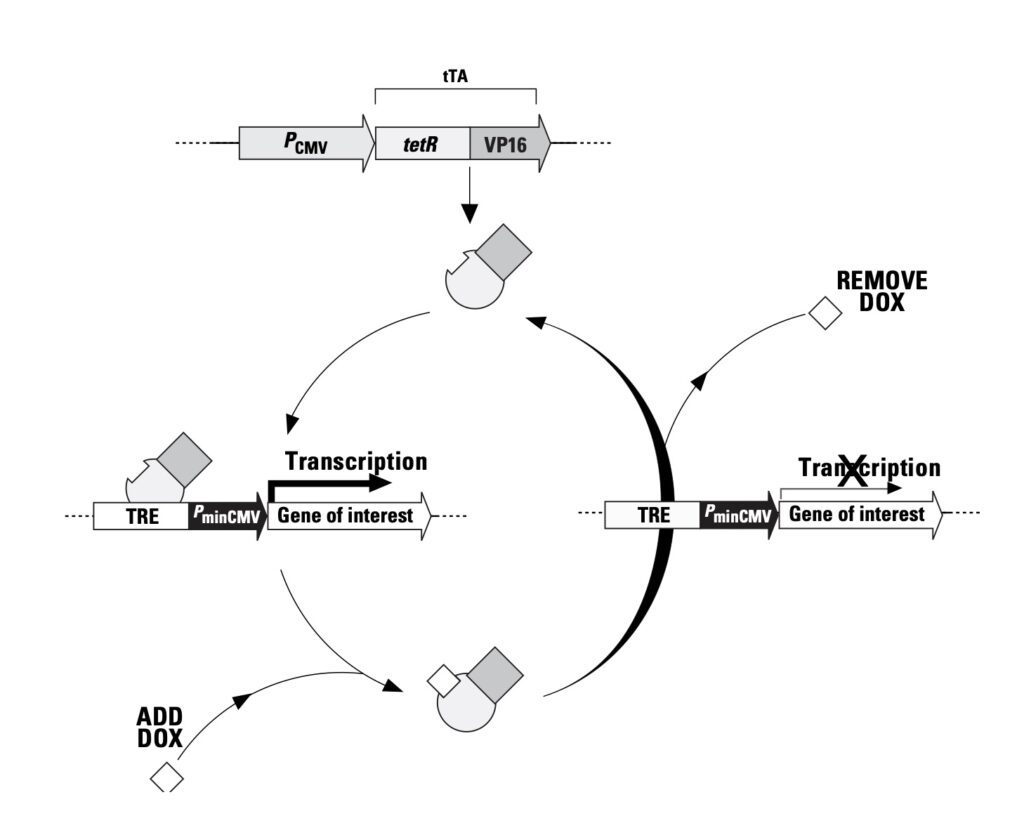
Come vediamo in figura 4 (da leggere in senso antiorario seguendo l’andamento delle frecce) il sistema Tet-Off prevede che tTA leghi la regione TRE e attivi la trascrizione del gene di interesse solo in assenza di tetraciclina (o doxiciclina). Se invece è presente si lega a tTA inducendone una modifica conformazionale impedendole di legare la regione TRE e bloccando così la trascrizione.
Tet-Off si preferisce generalmente quando vogliamo che il gene di interesse rimanga per lo più acceso. Infatti non sarebbe una condizione favorevole per le cellule rimanere troppo tempo in un ambiente in presenza di tetraciclina. Per questo motivo ci si limita a inserirla solo in periodi limitati di tempo per spengere il gene di interesse, che spesso è necessario alla vitalità cellulare.
Sistema Tet-on
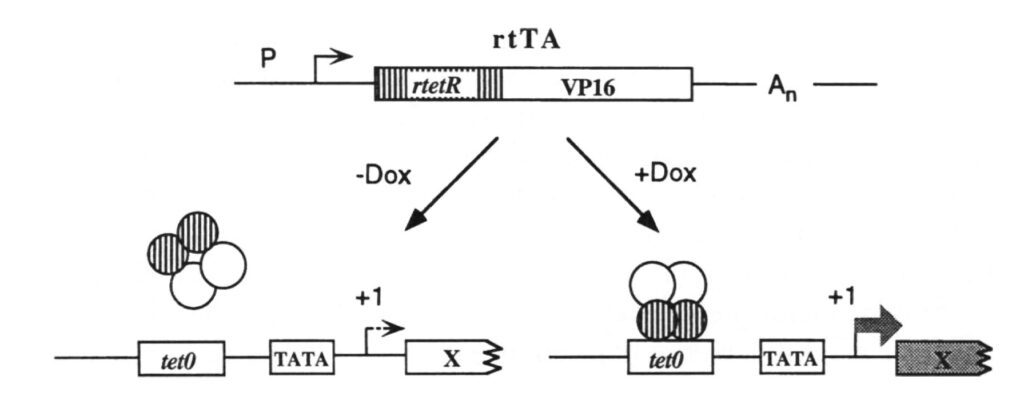
Il sistema Tet-On è simile a Tet-Off con la differenza che la proteina regolatrice non è tetR bensì r-tetR dove la “r” sta per “reverse” indicando che risponde in modo opposto alle condizioni di presenza o assenza della doxiciclina (in questo caso non è tetraciclina dipendente). r-tetR a troviamo sempre come proteina di fusione con VP16 formando la proteina r-tTA (r sta ancora per reverse). In questo modo, come si vede in figura 5, il transattivatore rtTA legherà il dominio TRE (e attiverà la trascrizione del gene di interesse) soltanto in presenza di doxiciclina. A differenza del sistema Tet-Off, Tet-On si preferisce usare quando vogliamo che il gene target rimanga per lo più spento, perchè per esempio traduce per un prodotto citotossico. Anche in questo caso il motivo è che così facendo la doxiciclina si aggiunge per periodi brevi e soltanto quando vogliamo attivare il gene.
Vantaggi del sistema Tet
- Riduzione al minimo di attività di background di espressione del gene di interesse da parte del promotore di CMV. Infatti si attiva solo in presenza o assenza di tetraciclina o doxiciclina, in base che si usi Tet-On o Tet-Off.
- Attività non pleiotropica, ovvero il sistema si basa su un meccanismo naturalmente presente nelle cellule procariotiche e non nelle eucariotiche. Per questo si ha la certezza che in cellule di mammifero la presenza o assenza di tetraciclina o doxiciclina regoli esclusivamente la trascrizione del nostro gene di interesse e non altri.
- Alta velocità di risposta, potendo infatti riscontrare effetti già a 30 minuti dall’aggiunta dell’antibiotico.
- Espressione massimale del gene di interesse è molto più alta rispetto a quella ottenibile con la presenza del solo promotore CMV.
- Espressione dose dipendente, potendo infatti controllare i livelli di espressione genica in base alla dose somministrata di antibiotico. Inoltre, le dosi di farmaco alle quali il sistema funziona sono ben al di sotto delle dosi citotossiche per le cellule in coltura.
- Semplicità e plasticità di realizzazione, infatti la trasfezione di vettori plasmidici è molto più semplice di altri modelli, soprattutto mediati da vettori virali. Inoltre il plasmide può raggiungere circa 30 mila basi e considerando che l’intero sistema Tet occupa soltanto 4,6 mila basi, c’è un consistente spazio per il nostro gene di interesse che può perciò raggiungere dimensioni considerevoli.
Svantaggi del sistema Tet
- Il vettore plasmidico non si integra, ma rimane in forma episomiale, ovvero che rimane nel nucleo della cellula senza integrarsi nel genoma. Questo può portare a perdere il vettore in modo passivo durante le replicazioni cellulari per semplice diluizione.
- La trasfezione plasmidica è applicabile a un numero limitato di tipologie cellulari. In particolare cellule in mitosi sono più semplici da trasfettare rispetto a cellule quiescenti, così come linee cellulari primarie rispetto a cellule immortalizzate. Per questo cellule come neuroni o cellule beta-pancreatiche difficilmente sono utilizzabili. Infine è una metodica limitata ad applicazioni in vitro e non in vivo, almeno sull’uomo.
- Si può verificare alta eterogeneità di trasfezione. Infatti, a fronte di un’alta efficienza del meccanismo di “gene delivery“, è probabile che alcune cellule contengano tante copie del gene di interesse ad altre poche o nessuna, portando così a una grande variabilità di espressione.
Possibili utilizzi in vitro e in vivo
Applicazioni in vitro
Le tecnologie genetiche possono servire per ottenere da cellule di interesse un prodotto naturale di quella cellula, modificato o addirittura una proteina completamente nuova. Questo è necessario quando vogliamo ottenere proteine in larga quantità, per esempio a scopo terapeutico. Un esempio è l’ormone umano della crescita (Growth Hormon, GH) per il trattamento del nanismo ipofisario, la cui origine di estrazione un tempo era soltanto dalle ipofisi rimosse da soggetti deceduti. Ovviamente questo trattamento portava svariati limiti sia di tipo etico sia, soprattutto, dovuti alla possibile trasmissione di malattie. Nel tempo venne compresa l’importanza dell’utilizzo di batteri e altri microorganismi come una sorta di “fabbrica di peptidi su richiesta” potendo inserirvi un gene di nostro interesse (come il GH) e spingerne la produzione anche in grandi quantità. In questo modo si abbatte ogni rischio di trasmissione di malattie, ma è anche una metodica semplice, a basso costo e di alta qualità.
Tuttavia, l’espressione di proteine animali in microorganismi batterici o eucariotici come i lieviti ha forti limitazioni. In particolare le modificazioni post-traduzionali che subisce una proteina per arrivare al prodotto biologicamente attivo sono molto diverse da quelle umane e animali, rischiando di ottenere polipeptidi parzialmente attivi o addirittura totalmente inattivi. Per questo allora, soprattutto quando si parla di produzione di proteine animali e umane, vi è la necessità di utilizzare cellule di mammifero. Ovviamente ci sono vari tipi di clonaggio disponibile, di tipo plasmidico, virale, retrovirale e mediante vescicole. Tuttavia così come in certi casi si desidera un’espressione elevata e costitutiva, certe volte è desiderabile poterla modulare. Il sistema Tet-On Tet-Off rappresenta proprio uno dei metodi più utili e efficienti a tal scopo.
Applicazioni in vivo
In vivo è possibile realizzare modelli di topo transgenici tetraciclina inducibili. Questi sono realizzati quasi sempre partendo da linee genitoriali separate in cui una è geneticamente modificata per contenere le informazioni di tTA o rtTA, mentre la seconda contenente il gene di interesse sotto il controllo del promotore TRE. Dall’accoppiamento delle due linee derivano (nella generazione filiale) modelli che permettono di controllare mediante il tamoxifene (o doxiclina) i livelli di espressione del gene in analisi. Ovviamente il farmaco viene somministrato a dosi specifiche tramite l’alimentazione, o nella dieta solida o nell’acqua di abbeveraggio. Inoltre, mediante l’utilizzo combinato del sistema Tet-On Tet-Off con Cre-LoxP è anche possibile controllare non solo quanto gene far esprimere, ma anche dove. Possiamo quindi un’espressione del gene di interesse tetraciclina dipendente tessuto specifica.
Fonti
- https://www.takarabio.com/documents/User%20Manual/PT3001/PT3001-1_100912.pdf
- Das AT, Tenenbaum L, Berkhout B. Tet-On Systems For Doxycycline-inducible Gene Expression. Curr Gene Ther. 2016;16(3):156-167. doi:10.2174/1566523216666160524144041
- Gossen M, Bujard H. Tight control of gene expression in mammalian cells by tetracycline-responsive promoters. Proc Natl Acad Sci U S A. 1992 Jun 15;89(12):5547-51. doi: 10.1073/pnas.89.12.5547. PMID: 1319065; PMCID: PMC49329.
- https://en.vectorbuilder.com/resources/vector-system/pRP_Tet_On.html
- Saunders TL. Inducible transgenic mouse models. Methods Mol Biol. 2011;693:103-15. doi: 10.1007/978-1-60761-974-1_7. PMID: 21080277.
- Gossen M, Freundlieb S, Bender G, Muller G, Hillen W, Bujard H. Transcriptional activation by tetracyclines in mammalian cells. Science. 1995;268:1766–1769. doi: 10.1126/science.7792603.
- Jeremy W. Dale, Malcolm Von Schantz, Plant Nick; “Dai Geni ai Genomi – principi e applicazioni della tecnologia del DNA ricombinante” Terza edizione, EdiSES, ISBN 978-88-7959-783-8
- https://youtu.be/42FPB2joEHE
Crediti immagini:
- Immagine in evidenza: https://www.osservatorioterapieavanzate.it/terapie-avanzate/terapia-genica/terapia-genica-obiettivo-mucopolisaccaridosi
- Figura 1: Til, Niek & Markusic, David & Rijt, Roos & Kunne, Cindy & Hiralall, Johan & Vreeling, Heleen & Frederiks, Wilma & Oude Elferink, Ronald & Seppen, Jurgen. (2005). Kupffer Cells and Not Liver Sinusoidal Endothelial Cells Prevent Lentiviral Transduction of Hepatocytes. Molecular therapy : the journal of the American Society of Gene Therapy. 11. 26-34. 10.1016/j.ymthe.2004.09.012.
- Figura 2, 3 e 4: https://www.takarabio.com/documents/User%20Manual/PT3001/PT3001-1_100912.pdf
- Figura 5: Gossen M, Freundlieb S, Bender G, Muller G, Hillen W, Bujard H. Transcriptional activation by tetracyclines in mammalian cells. Science. 1995;268:1766–1769. doi: 10.1126/science.7792603.