Introduzione al DNA
Il DNA è una acido nucleico, scoperto per la prima volta attorno al 1870, quando il chimico svizzero Friederich Miescher isolò dai globuli bianchi una sostanza ricca di fosforo cui diede il nome di nucleina. La nucleina venne studiata fino ad elaborarne le proprietà chimiche portando alla scoperta del DNA.
Inizialmente, infatti, le sue funzioni erano sconosciute. Era chiara la sua composizione: basi azotate, gruppi fosfato e zuccheri pentosi. Il monomero della molecola, composto da una base azotata, uno zucchero pentoso (deossiribosio) e uno o più gruppi fosfato, venne nominato “nucleotide”.
Oggi sappiamo che il DNA è alla base della vita e che rappresenta un “codice” che, una volta processato, è in grado di fornire istruzioni per la sintesi proteica.
Per arrivare a queste conclusioni, tuttavia, furono necessari una serie di esperimenti, che provarono la sua funzione.
Il principio trasformante di Griffith
Nel 1928, il microbiologo inglese Frederick Griffith stava studiando la polmonite. In particolare, egli cercava un vaccino che potesse scongiurare la possibilità di contrarre la malattia. Per i suoi studi, decise di utilizzare Streptococcus pneumoniae, e nello specifico due ceppi: il ceppo S ed il ceppo R. Il primo ceppo era liscio (da cui S di smooth) e risultava patogeno per i topi, causava polmonite ed infine la morte. Il secondo era definito “rugoso” ed era invece benigno ed i topi inoculati con tale ceppo rimanevano in vita. La differenza tra i due ceppi era data proprio dal fenotipo: mentre il primo possedeva una capsula polisaccaridica che lo rendeva invisibile al sistema immunitario, il secondo ne era sprovvisto, e dunque veniva facilmente neutralizzato dal sistema immunitario delle cavie.
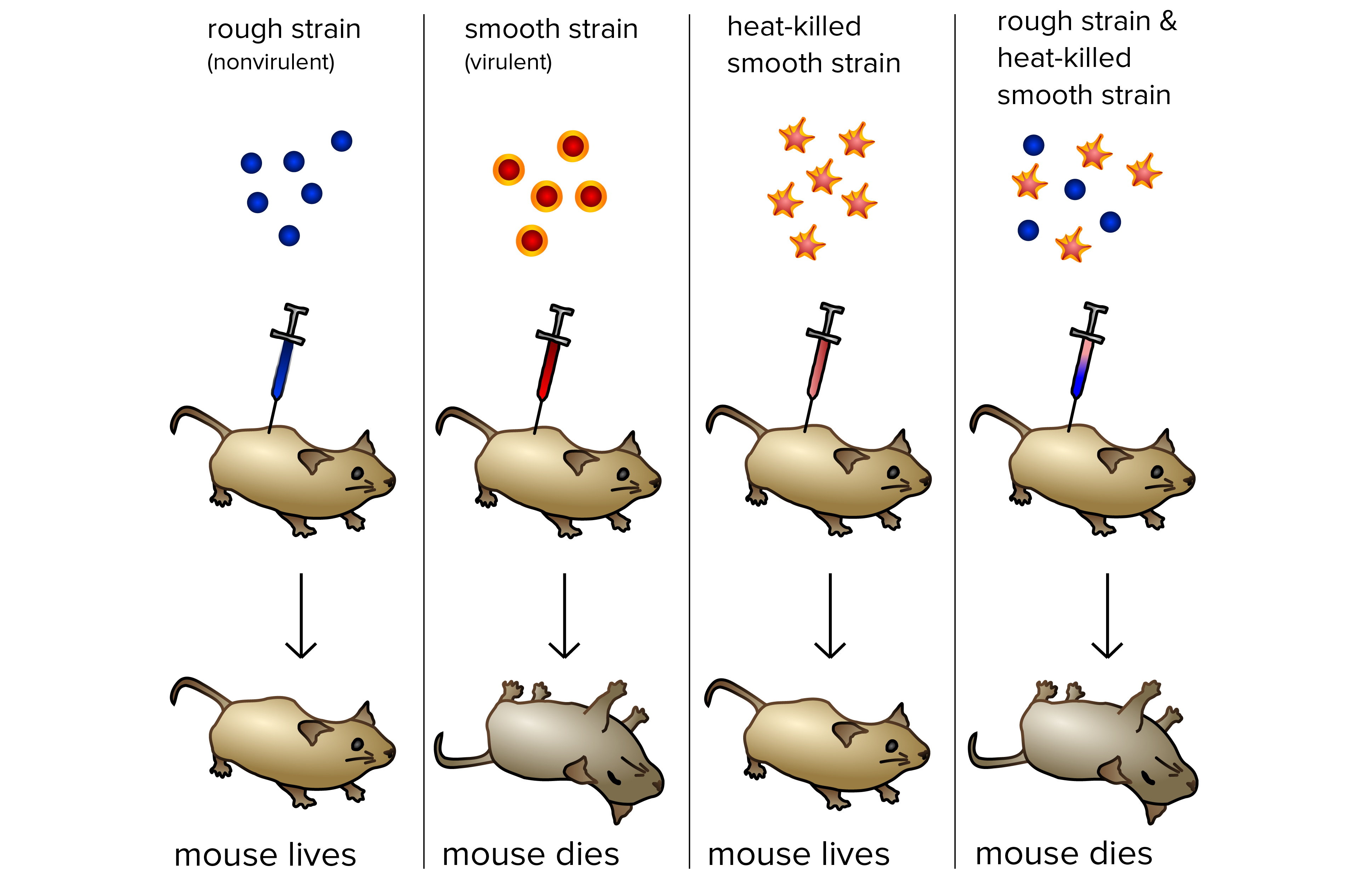
L’esperimento continuò con l’inoculo del ceppo S, stavolta in seguito alla sua inattivazione (alzando la temperatura della soluzione prima dell’iniezione) e si osservò che dopo tale trattamento, il ceppo perdeva l’aspetto patogeno. Se però il ceppo inattivato patogeno si iniettava insieme al ceppo R benigno vitale, si assisteva alla morte di tutte le cavie.
In seguito a questo esperimento, Griffith ipotizzò che qualcosa del ceppo S riusciva a penetrare nel ceppo R, trasformandolo in un ceppo patogeno. Questo elemento venne denominato, proprio per le sue proprietà, “principio trasformante”.
La “bomba” di Avery
Nel 1944 Oswald T. Avery riprese l’ipotesi del principio trasformante di Griffith, cercando di identificarlo. Egli mise in diverse provette dei batteri vitali del ceppo S, aggiungendo in ognuna dei reagenti specifici a seconda della classe molecolare. Utilizzò, infatti, tripsina per degradare le proteine, DNAsi per digerire il DNA e così via, ottenendo in ogni provetta una sola macromolecola proveniente dal ceppo originario patogeno S. Infine, inoculò nei topi il contenuto di ciascuna provetta insieme al ceppo R vitale.
I risultati furono chiari: solo nel caso in cui la molecola intatta era il DNA il ceppo R diventava patogeno e passava da rugoso a liscio, uccidendo l’animale. Ciò portò alla convinzione che il “principio trasformante” fosse proprio il DNA che, passando dal ceppo S al ceppo R, modificava la natura del batterio, rendendolo patogeno.

Questa scoperta ebbe un enorme impatto nel mondo scientifico, motivo per cui si denominò “bomba di Avery”.
L’esperimento del frullatore di Hershey e Chase
Nel 1952 Alfred Hershey e Martha Chase utilizzarono i batteriofagi per confermare le ipotesi formulate nel corso degli anni riguardo alla natura del DNA come materiale genetico. Utilizzarono i fagi per questo esperimento, poiché questi piccoli virus sono molto semplici: consistono infatti solamente in capside (di origine proteica) e materiale genetico. Quando un fago infetta una cellula, infatti, il materiale genetico viene spinto all’interno, mentre il capside rimane fuori.
I due scienziati marcarono radioattivamente il DNA del fago con del fosforo radioattivo (32P) e le proteine capsidiche con zolfo radioattivo (35S). Subito dopo l’infezione, posero il brodo di coltura in un frullatore, che provocava il distacco del capside vuoto dai batteri. In seguito, si centrifugava il brodo, per poi andare a verificare che il DNA marcato fosse all’interno dei batteri, mentre le proteine marcate all’esterno.
Ottenendo il risultato sperato, Hershey e Chase furono gli ultimi di una lunga catena di scienziati che riuscirono a dimostrare non solo che il “principio trasformante” era il DNA, ma che quest’ultimo rappresentava il materiale genetico delle cellule.
Struttura chimica del DNA
Il DNA è costituito da catene polinucleotidiche, ossia polimeri lineari costituiti da monomeri detti “nucleotidi”. Il nucleotide è composto da tre parti fondamentali: una base azotata, uno zucchero pentoso (nel caso del DNA il deossiribosio) e uno o più gruppi fosfato. Le basi azotate possono essere classificate in due tipologie: purine (adenina e guanina, costituite da due anelli eterociclici) e pirimidine (timina e citosina, formate da un solo anello eterociclico).
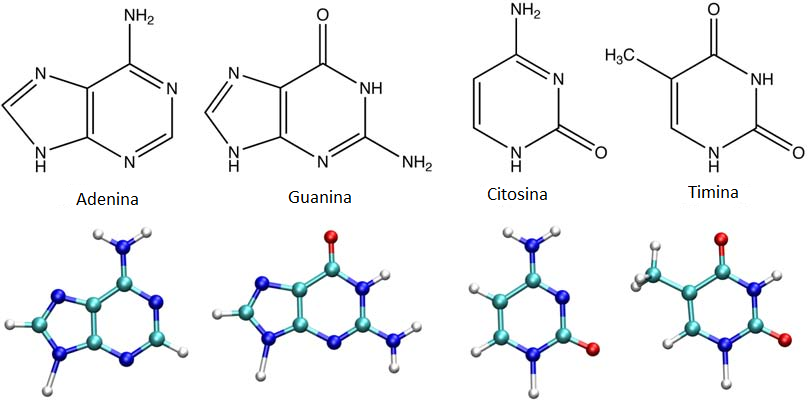
Le purine e le pirimidine si accoppiano tra loro in modo preciso, ovvero l’adenina si appaia alla timina e la guanina alla citosina. Qualsiasi mal appaiamento, costituendo una mutazione, è corretto dai meccanismi di riparo del DNA. Il legame tra adenina e timina consiste in due legami a idrogeno, mentre quello tra citosina e guanina in tre, motivo per cui è più facile aprire una molecola di DNA ricca in AT ed è invece più complesso nel caso di un tratto di DNA contenente molte CG.
La base azotata si lega allo zucchero in posizione 1′ tramite un legame glicosidico che interessa l’N1 delle pirimidine e l’N9 delle purine.
I nucleotidi sono invece legati tra loro dal legame fosfodiesterico, che comprende due legami esteri: uno tra un gruppo fosfato del nucleotide precedente e il carbonio 5′ dello zucchero e uno tra il carbonio 3′ dello zucchero e il nucleotide successivo.
Tale legame conferisce asimmetria alla catena, non strutturale, bensì di carica.
Struttura fisica del DNA: la doppia elica di Watson e Crick e gli istoni
Nel 1953 James Watson e Francis Crick scoprirono la struttura secondaria a doppia elica del DNA. Secondo il modello proposto, le basi, piatte, sono poste quasi perpendicolarmente allo scheletro zucchero-fosfato, formando una serie di gradini in una scala a chiocciola. Le due catene laterali, costituite da zucchero e gruppi fosfato, si definiscono antiparallele, poiché scorrono in direzioni opposte.
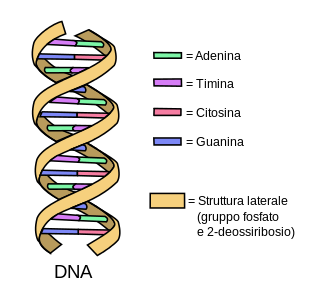
L’elica del DNA presenta un’asimmetria strutturale dovuta al posizionamento esterno del deossiribosio, e ciò causa la presenza di due tipologie di solchi: il solco minore e il solco maggiore.
Il solco maggiore, essendo più esposto, costituirà la base di ancoraggio di proteine specifiche che interagiscono con il DNA.
La presenza di gruppi fosfato polarizza la molecola all’esterno. Tale polarizzazione è funzionale: non solo consente alle molecole di DNA di trovarsi in ambienti acquosi senza dissipare energie, ma gli consente di legarsi a strutture proteiche dette istoni che permetteranno un’ulteriore condensazione della molecola. Il complesso molecola di DNA-istoni è detto nucleosoma.
…Gli istoni
Ogni cellula umana, infatti, possiede circa 2 metri di DNA, che dovranno essere opportunatamente ripiegati per far sì che rientrino nel nucleo. A tale scopo delle proteine dette istoni si associano e, grazie alla polarità positiva delle loro code proteiche, legano la doppia elica. Gli istoni dunque la arrotolano attorno a sé, comprimendo ulteriormente la lunghissima molecola. Tali proteine, presenti negli eucarioti e in forma simile nei procarioti, sono responsabili della formazione della cromatina, ossia una molecola di DNA estremamente condensata.
La condensazione del DNA non è solo funzionale da un punto di vista puramente spaziale, ma lo è anche da un punto di vista genetico: più la molecola è condensata, meno sarà accessibile. Ciò comporta la possibilità della cellula di “spegnere” e “riattivare” sequenze geniche. Esse possono essere sia molto corte (come nel caso di un singolo gene che non è necessario codificare), sia molto lunghe (come il secondo cromosoma X femminile). Tale abilità è studiata e approfondita dall’epigenetica.
I codoni: l’unità organizzativa del codice genetico
Il codice genetico ha un’organizzazione molto precisa. Essa consente di codificare proteine funzionali, ma solo nel caso in cui tutte le condizioni di conservazione siano rispettate.
Il codice genetico è organizzato in codoni. Ogni codone è formato dalla sequenza di tre basi azotate in un ordine preciso. Ad ogni codone, infine, corrisponde un amminoacido.
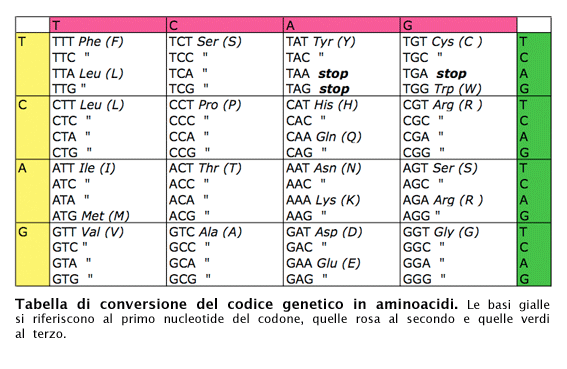
Va da sé che è molto importante mantenere l’integrità del codone, soprattutto bisogna evitare l’inserimento di una base o la delezione di una base. Questa mutazione, apparentemente innocua, causerebbe invece un errore di lettura del frameshift, ossia della “finestra di lettura” del codice genetico. Slittando di una base, tutti gli appaiamenti e tutti i codoni risulteranno corrotti e porteranno alla formazione di una proteina aberrante, ossia non funzionante.
Inizialmente infatti, “rilegge” il trascritto tramite la DNA polimerasi stessa, che non solo funziona come enzima replicativo, ma anche come riparatore di errori in prima battuta. L’abilità aggiuntiva della polimerasi si chiama proofreading ed è sempre attiva. Ciò diminuisce esponenzialmente la percentuale di errori replicativi.
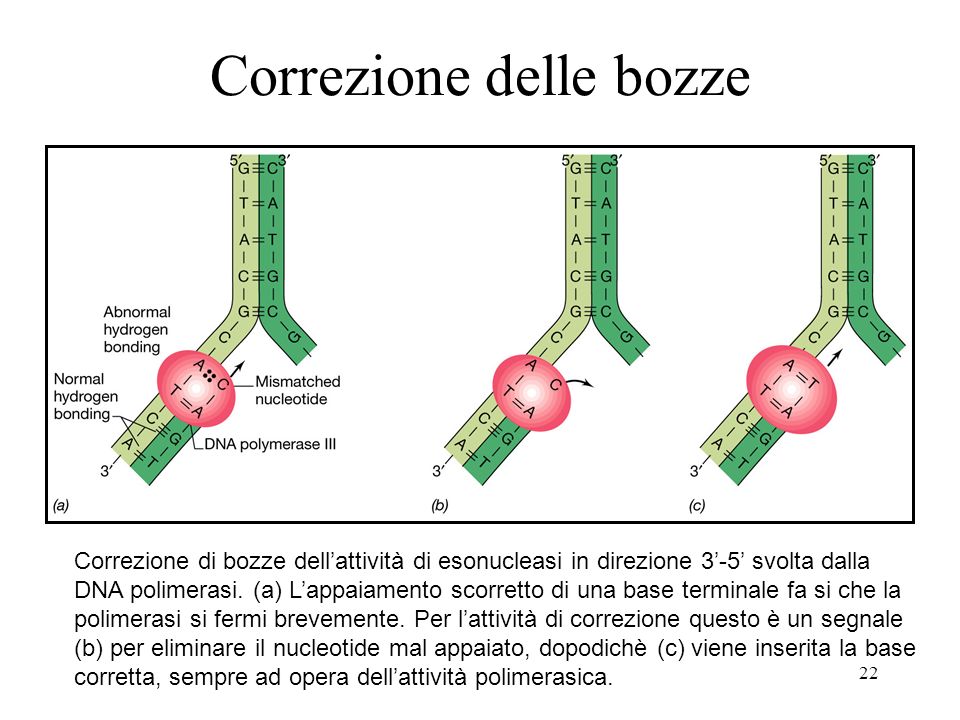
E’ estremamente importante che l’integrità del codice genetico venga mantenuta per il corretto funzionamento della sintesi proteica e dunque della vita stessa. Per questo motivo, il DNA è in grado di “proteggersi” da eventuali mutazioni ed errori su più livelli.
Esistono molte altre modalità contemporanee e/o successive alla replicazione che la cellula attua per diminuire le mutazioni, come ad esempio meccanismi di escissione del nucleotide o di una porzione intera di DNA, che consentono al codice genetico di mantenere la sua integrità.
Francesca Buratti
Fonti
- Biologia molecolare della cellula – B. Alberts
- Biologia molecolare – F. Amaldi
1 commento su “Il DNA, acido nucleico alla base della vita – scheda didattica”