Il microbiota intestinale
Il nostro tratto gastro-intestinale, oltre alla pelle, la bocca, la vagina e le vie respiratorie, è abitato da comunità microbiche particolari con strutture e funzioni specifiche. Per microbiota intestinale (Fig.1) si intende l’ecosistema complessivo formato da funghi, virus e batteri che si sono adattati a vivere sulla superficie mucosa dell’intestino o nel suo lume, sviluppandosi immediatamente dopo la nascita, influenzato dalla modalità del parto, dalla nutrizione iniziale e dal genotipo dell’ospite.
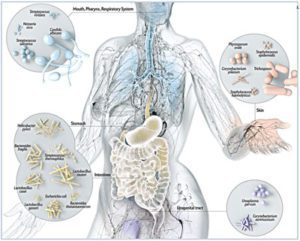
Composizione del microbiota: la nostra “impronta digitale” microbica
Nel corso dei primi 3 anni di vita, l’iniziale diversità microbica si normalizza e tende a restare tale. Infatti, nell’adulto la composizione del microbiota tende a essere stabile in condizioni fisiologiche in quanto modulata da diversi fattori, fra cui l’età, l’assunzione di antibiotici e la dieta. L’analisi delle comunità microbiche intestinali ha definito la presenza di tre varianti predominanti rappresentati da Bacteroides, Prevotella e Ruminococcus. Ognuno di noi ha un proprio profilo di specie diverso da quello di altri individui, ma anche una componente di almeno 57 specie comuni a tutti gli individui per i quali i phyla dominanti sono solo due, i Bacteroidetes e i Firmicutes. Questi due phyla costituiscono più del 90% delle categorie filogenetiche. Le restanti sono rappresentate da Bacteroidetes, Dorea/Eubacterium/Ruminococcus come pure Bifidobacteria, Proteobacteria, Streptococchi e Lactobacilli.
La normale interazione fra microbi intestinali e il loro ospite umano è un rapporto simbiotico, benefico per entrambi. L’ospite mette a disposizione un habitat ricco di nutrienti e il microbiota conferisce elementi utili alla sua salute. L’insieme dei geni dei batteri viene invece definito microbioma.
Tecniche di studio del microbiota
Le tecniche di laboratorio coltura-indipendenti hanno consentito la caratterizzazione delle comunità microbiche intestinali, migliorando la capacità di identificare le singole specie di microrganismi e le loro quantità in campioni di feci (Fig. 2).

La maggior parte degli studi molecolari è stata condotta analizzando la sequenza del gene che codifica per la subunità 16S dell’RNA ribosomiale (rRNA). I tre approcci più comunemente utilizzati per studiare la diversità del microbiota intestinale sono il targeting genico mediante la reazione a catena della polimerasi (PCR), le tecniche di ibridazione basate su sonde oligonucleotidiche fluorescenti, come l’ibridazione fluorescente in situ (FISH), e le tecniche di fingerprinting molecolare, come la denaturazione elettroforetica su gel a gradiente (DGGE).
Le tecniche di laboratorio coltura-indipendenti come la PCR associata all’uso della trascrittasi inversa (RT-PCR), l’ibridazione fluorescente in situ dell’rRNA (FISH), l’elettroforesi su gel in gradiente denaturante o di temperatura (DGGE e TGGE) e la tecnologia “microarray” forniscono sempre più informazioni sulla composizione, i determinanti e i ruoli funzionali del microbiota umano.
Le tecniche di targeting genico utilizzano primer gene-specifici per amplificare i geni bersaglio come le regioni conservate del 16S rRNA. Questo approccio è stato ampiamente applicato all’analisi del microbiota intestinale, rivelando la sostanziale diversità batterica e studiando gruppi di microrganismi difficilmente coltivabili. Inoltre, la tecnica della PCR associata all’uso della trascrittasi inversa (RT-PCR) viene utilizzata per clonare i geni batterici a partire da campioni di feci. Un altro metodo per analizzare il microbiota intestinale è l’ibridazione fluorescente in-situ dell’rRNA (FISH). Basate sulle sequenze dell’rRNA 16S, le sonde possono essere sintetizzate in modo tale da legarsi specificamente ai geni degli organismi di interesse. Utilizzando sonde con marcatori fluorescenti diversi, possono essere visualizzati simultaneamente all’interno di comunità complesse diversi tipi di microrganismi.
Le tecniche di “fingerprinting” molecolare di solito vengono utilizzate per analizzare la diversità microbica. La DGGE (elettroforesi su gel in gradiente denaturante) e la TGGE (elettroforesi su gel a gradiente di temperatura) sono forse le tecniche più comunemente utilizzate e sono utili nel fornire una panoramica della diversità del microbiota intestinale. Queste tecniche hanno rivelato una significativa diversità microbica nei campioni di feci, ma forniscono informazioni piuttosto limitate e il recente sviluppo delle piattaforme metagenomiche sta rapidamente sostituendo queste tecniche.
La metagenomica esplora la composizione globale delle comunità microbiche, combinando la biologia molecolare e le tecniche genetiche per identificare e caratterizzare il materiale genetico da ambienti microbici complessi. La tecnologia “microarray” è un potente strumento per la rapida individuazione di migliaia di sequenze geniche dell’rRNA 16S, fornendo un profilo delle comunità microbiche presenti nell’intestino umano.
Giusi Balzano
Fonti:
- Gill SR, Pop M, Deboy RT, et al. Metagenomic analysis of the human distal gut microbiome. Science 2006; 312:1355-9.
- Kamada N, Chen GY, Inohara N, Núñez G. Control of pathogens and pathobionts by the gut microbiota. Nat Immunol 2013; 14: 685-90.
- Lozupone CA, Stombaugh JI, Gordon JI, Jansson JK, Knight R. Diversity, stability and resilience of the humangut microbiota. Nature 2012; 489: 220-30.
- Faith JJ, Guruge JL, Charbonneau M, et al. The long-term stability of the human gut microbiota. Science 2013; 341:84-7.
- Arumugam M, Raes J, Pelletier E, et al. Enterotypes of the human microbiome. Nature 2011; 473: 174-80.
- Su C, Lei L, Duan Y, Zhang K-Q, Yang J. Culture-independent methods for studying environmental microorganisms: methods, application, and perspective. Appl. Microbiol. Biotechnol. 2011.
- Karlsson CLJ, Molin G, Cilio CM, Ahrné S. The pioneer gut microbiota in human neonates vaginally born at term-a pilot study. Pediatr. Res. 2011; 70:282-6.
- Swidsinski A, Loening-Baucke V, Herber A. Mucosal flora in Crohn’s disease and ulcerative colitis – an overview. J. Physiol. Pharmacol. 2009; 60 Suppl 6:61-71.