Klebsiella (Figura 1) è una delle principali cause di infezione ospedaliera a livello globale. Come molte altre specie batteriche è in grado di acquisire resistenza a più farmaci contemporaneamente. Questa multi-farmacoresistenza ha reso necessario avviare politiche di sorveglianza mirate al controllo della diffusione di questo batterio. Esse sono basate in primis sull’identificazione delle nicchie e delle sorgenti di infezione.
Klebsiella però è molto difficile da debellare e questo si deve a due requisiti, in realtà comuni a molte specie batteriche:
- la possibilità di poter trasferire materiale genico mobile, ad esempio plasmidi, a cellule non-figlie (TGO o trasmissione genica orizzontale);
- la presenza di un gene specifico di resistenza (riscontrabile in ceppi di K. pneumoniae) detto betalattamasi che rende poco o per nulla efficaci l’azione degli antibiotici beta-lattamici;
Anche per questo l’approccio gene-centrico del seguente studio, condotto nell’ambito del progetto SPARK, fornisce un mezzo molto utile.
Figura 1 – Immagine al microscopio di Klebsiella . La figura è rappresentativa di Klebsiella pneumoniae, la specie più diffusa: si evince la morfologia a bastoncello e l’assenza di strutture flagellari. Le infezioni da Klebsiella sono contratte principalmente in ambiente ospedaliero ed interessano tessuti specifici e delicati, più facilmente esposti ad infezioni di diversa natura: un esempio sono polmoniti ed infezioni del tratto urinario (soprattutto a causa dei cateteri).
Il progetto SPARK
SPARK, acronimo di Spread of Population-wide Antibiotic Resistance in Klebsiella, guidato dall’Università di Bath (GB) e finanziato dall’Unione Europea, è un progetto internazionale che vede coinvolte diverse realtà universitarie e non.
Il progetto ha il fine ultimo di studiare l’antibiotico-resistenza di ogni ceppo di Klebsiella per elaborare innovative strategie di controllo ed evitare l’insorgenza di nuove infezioni e focolai epidemici.
L’unico team italiano partecipante è rappresentato dal gruppo di ricerca dell’Università degli Studi di Pavia, in collaborazione con il Policlinico San Matteo e l’Istituto Zooprofilattico, che ha contribuito alla caratterizzazione genetica del “super-resistente batterio” K. pneumoniae sul territorio di Pavia.
I batteri del genere Klebsiella
Il genere Klebsiella, afferente alla famiglia degli Enterobacteriaceae, si compone di batteri bastoncellari, capsulati e Gram-negativi, asporigeni ed incapaci di muoversi. Questi batteri sono per natura ubiquitari: si ritrovano facilmente sia nelle acque che nel suolo e nelle piante. Possono comportarsi come specie commensale nell’intestino degli animali, difatti sono normalmente presenti nell’intestino dell’uomo singolarmente o formanti catenelle.
I batteri appartenenti a questo genere non sono di per sé patogeni: K. pneumoniae e Klebsiella oxytoca, ad esempio, colonizzano l’intestino di molti vertebrati e non causano alcun disturbo. Proprio queste due specie però, in un soggetto delicato e predisposto, determinano infezioni nosocomiali anche molto difficili da debellare: si parla di multi-farmacoresistenza.
Klebsiella oxytoca
K. oxytoca è la seconda specie potenzialmente patogena di Klebsiella più comune nell’uomo, dopo K. pneumoniae. Viene suddivisa in ben 6 linee filogenetiche: Ko1, Ko2, Ko3, Ko4, Ko6, e Ko7.
Soltanto di recente, studi condotti all’Università degli Studi di Pavia hanno permesso di comprendere che le due sottospecie classificate come Ko3 e Ko4, sono in realtà due specie nuove afferenti allo stesso genere. Ko3 e Ko4, sulla base delle caratteristiche genomiche, fenotipiche e proteomiche, condividono lo stesso progenitore di K. oxytoca ma nonostante gli attributi molto simili, le differenze presenti hanno reso necessario un nuovo lavoro di classificazione tassonomica (Figura 2).
Si è proposto di nominare K. spallanzanii e K. pasteurii, rispettivamente, le classi filogenetiche fino ad oggi conosciute come Ko3 e Ko4.
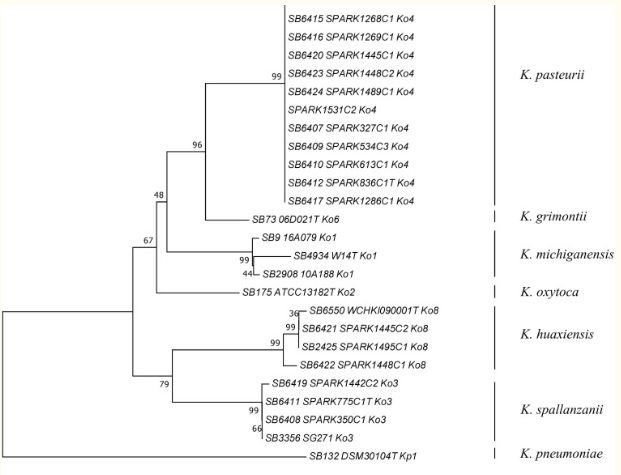
Figura 2 – Albero filogenetico di Klebsiella. Questa classificazione, che sottolinea la presenza di K. pasteurii e K. spallanzanii, è basata sulle sequenze concatenate dei geni gyrA e rpoB, che da soli consentirebbero un’identificazione affidabile; tuttavia gli isolamenti su terreni specifici, l’analisi della sequenza genomica, la spettrometria di massa (MALDI-ToF) e l’uso di biomarcatori filogenetici hanno dato un’ulteriore conferma permettendo così la definizione delle due nuove specie batteriche.
Klebsiella pasteurii
Dalle prime analisi K. pasteurii è risultato presente solo nel materiale fecale di origine umana e bovina. Il batterio è così denominato in onore del microbiologo francese Louis Pasteur, padre della moderna microbiologia e di scoperte salva-vita nel campo delle malattie infettive, vaccini e pastorizzazione,
L’identificazione preliminare basata sulle caratteristiche morfologiche ha rivelato che K. pasteurii è un bacillo Gram-negativo dotato di una capsula esterna formata per la maggioranza da polisaccaridi ma non di appendici cellulari quali i flagelli che ne consentirebbero il movimento. Al microscopio appare dunque non mobile e non formante spore, in grado di modellarsi in grandi colonie rotondeggianti, biancastre e lucenti. Ai test biochimici si è mostrato:
- indolo-positivo: il batterio è in grado di metabolizzare il triptofano essendo l’indolo un sottoprodotto derivante dalla digestione di questo amminoacido;
- ureasi-negativo;
- lattosio fermentante: positivo all’O.N.P.G. Test per la determinazione della presenza dell’enzima β-galattosidasi;
- Voges-Proskauer test-positivo: ossida il glucosio attraverso una fermentazione glicol-butilenica producendo, principalmente etanolo, 2,3 butandiolo e acido formico;
- lisina decarbossilasi positivo (a differenza di K. aeruginosa);
- ornitina decarbossilasi negativo;
Inoltre, a differenza degli altri ceppi di K. oxytoca, questo batterio ha la capacità di fermentare l’acido gliossilico e lo zucchero D-melezitosio ma non l’acido α-chetoglutarico (Figura 3).
Klebsiella spallanzanii
K. spallanzanii è stato isolato dalle urine umane e dalle feci bovine e riscontrato per questo anche su terreni ad uso agricolo. Il nuovo ceppo prende il nome dal biologo italiano Lazzaro Spallanzani, vissuto nel XVIII secolo e ricordato soprattutto per i suoi studi sperimentali sulle funzioni corporee. Grazie ai suoi esperimenti sull’apparato respiratorio e digestivo, scoprì le modalità di secrezione del succo gastrico. Egli, inoltre, si occupò di riproduzione animale e diede un grosso contributo alla causa ottenendo, già all’epoca, la prima fecondazione artificiale con uova di anfibi (rane e rospi).
Come il precedente, K. spallanzanii è un batterio Gram-negativo, di forma bastoncellare e asporigeno, con capsula e non dotato di motilità. Le caratteristiche generali e le attività enzimatiche specifiche sono identiche a quelle delle restanti specie del genere Klebsiella: trattasi dunque di un batterio formante colonie, in grado di produrre β-galattosidasi ed indolo; inoltre si registra la presenza dell’enzima lisina decarbossilasi ma l’ODC-test è invece negativo.
Si differenzia però dalle altre sotto-specie del complesso K. oxytoca per essere ureasi-positivo. Il batterio inoltre è incapace di utilizzare molecole organiche osmolite come L-prolina.
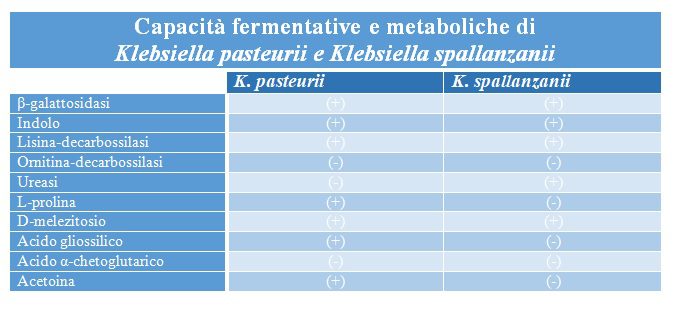
Figura 3 – Caratteristiche metaboliche di K. pasteurii e K. spallanzanii. Su colonie isolate sono stati effettuati test biochimici specifici: i nuovi ceppi batterici fermentano mediamente gli zuccheri lattosio e D-melezitosio e sono dotati di enzimi specifici per la decarbossilazione degli amminoacidi triptofano e lisina. Manca invece la capacità di digerire l’ornitina (ODC-test negativo). K. spallanzanii produce l’enzima ureasi, che scinde l’urea in NH3, per tamponare l’acidità del pH mentre K. pasteurii è in grado di incorporare L-prolina. Entrambi i ceppi batterici digeriscono l’acido α-chetoglutarato ma solo K. pasteurii metabolizza anche l’acido gliossilico. La reazione Voges-Proskauer ha confermato una fermentazione butandiolica (produzione di acetoina durante la fermentazione del glucosio) per K. pasteurii.
Conclusioni dello studio
Dalle analisi biochimiche per l’identificazione delle nuove specie risultano differenze, seppur minime, soprattutto per quanto concerne le capacità fermentative e metaboliche.
Grazie a questo progetto è stato possibile implementare le conoscenze sulle sequenze genomiche di Klebsiella e definire un pan-genome, ovvero un database che raggruppa geni presenti in tutti i ceppi all’interno del clade (genoma centrale) e geni presenti solo in alcuni ceppi e/o di geni specifici del ceppo (genoma accessorio o variabile).
I dati raccolti potranno essere utili per ricerche genetiche future.
Carla Caianiello
Fonti:
- Merla C., Rodrigues C., Passet V., Corbella M., Thorpe H.A., Kallonen T.V.S., Zong Z., Marone P., Bandi C., Sassera D.,10, Corander J., Feil E., J., Brisse S. (2019). Description of Klebsiella spallanzanii sp. nov. and of Klebsiella pasteurii sp. nov. Front. Microbiol. Doi: https://doi.org/10.3389/fmicb.2019.02360
- http://epidemiologiagenomica.sanmatteo.org/site/home/progetti/articolo11003880.html
- http://news.unipv.it/?p=43759
- https://www.microbiologiaitalia.it/batteriologia/klebsiella-pneumoniae/
- https://www.jpiamr.eu/wp-content/uploads/2016/11/SpARK.pdf
- https://www.studenti.it/glucosio.html
- https://www.tuasaude.com/bacteria-kpc-klebsiella-pneumoniae-carbapenemase/