Microbiota ed epigenetica
Negli anni recenti si è assistito ad un’esplosione di studi in due campi strettamente interconnessi: il microbiota e l’epigenetica.
Il network delle popolazioni microbiche, che albergano sulle superfici mucosali e cutanee, rappresenta il principale centro interno di segnalazione epigenetica dei sistemi biologici e questo evidenzia l’importanza dell’azione epigenetica svolta dal microbiota umano.
Microbiota ed epigenoma
Il tratto gastrointestinale umano ospita migliaia di miliardi di microrganismi commensali che compongono collettivamente il microbiota intestinale.
In altre parole, tali comunità microbiche sono emerse come una fonte critica di stimoli ambientali in grado di avere un impatto sulla salute e sulle malattie.
Tali comunità microbiche regolano l’omeostasi e l’alterazione nella loro composizione viene associata a diverse patologie, tra cui le malattie infiammatorie intestinali (IBD), le malattie cardiovascolari, le malattie metaboliche e perfino il cancro.
Tuttavia, i meccanismi cellulari e molecolari che riconoscono i segnali derivati da batteri commensali e che regolano l’omeostasi stanno iniziando ad essere chiariti solo di recente.
Modifiche epigenomiche altamente complesse consentono di alterare il programma trascrizionale di una cellula in risposta a segnali ambientali e cominciano così a fornire dati a favore di una correlazione tra microbiota e regolazione epigenetica.
Tali modifiche possono svolgere un ruolo chiave nella regolazione della relazione dinamica tra l’ospite e il suo microbiota.
In questo articolo vengono passati in rassegna alcune recenti evidenze per meglio comprendere l’azione epigenetica del microbiota e le vie epigenomiche che implicano un ruolo centrale delle deacetilasi istoniche (HDAC) nell’orchestrazione delle interazioni ospite-microbiota.
Meccanismi epigenetici
La ricerca scientifica evidenzia sempre più come i nostri comportamenti e le nostre scelte incidano sull’attivazione o sul silenziamento dei nostri geni.
Queste scoperte accostano al ruolo del DNA, finora considerato come unico determinante, quello dell’interazione con l’ambiente, in grado di attivare o disattivare i suoi componenti.
La regolazione trascrizionale dinamica è essenziale per mediare l’azione epigenetica del microbiota.
Le cellule eucariotiche impacchettano il loro DNA attorno alle proteine istoniche per formare una struttura di ordine superiore chiamata cromatina.
Le modificazioni epigenomiche alterano la struttura della cromatina e quindi alterano il programma trascrizionale di una cellula e tali modifiche sono mantenute dall’attività dinamica di vari enzimi modificanti e demodificanti, le cui attività possono essere influenzate dai metaboliti e da altri segnali ambientali.
In termini molecolari, i principali meccanismi di regolazione epigenetica della trascrizione sono i seguenti: modificazioni post-traduzionali delle code istoniche N-terminali, metilazione del DNA, codifica di varianti istoniche, rimodellamento della cromatina, attività di RNA non codificanti, ognuna delle quali può modificare l’espressione genica senza alterare la sequenza di DNA associata.
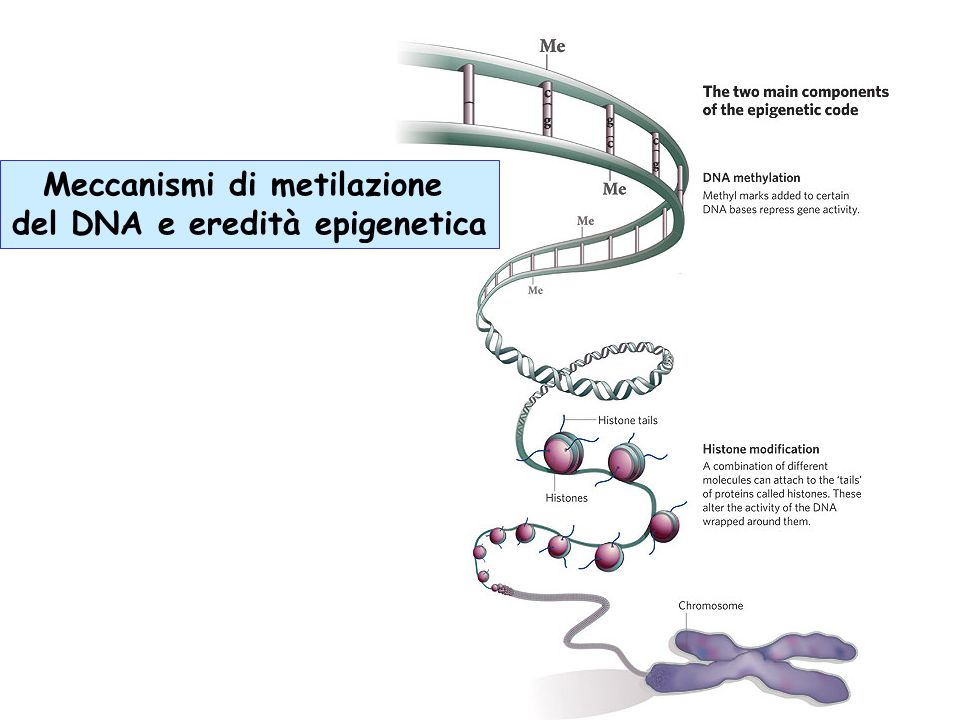
Le principali modifiche delle code istoniche sono l’acetilazione dei residui di lisina degli istoni H2A, H2B, H3 e H4 che permettono alla cromatina di assumere una conformazione chiamata eucromatina, meno condensata accessibile alle proteine (fattori di trascrizione) presenti sui promotori.
Al contrario, la deacetilazione degli istoni impacchetta la cromatina formando l’eterocromatina, che risulta inattiva.
La reazione di acetilazione è mediata dagli enzimi istone acetiltransferasi (HAT), mentre la deacetilazione dagli enzimi istone deacetilasi (HDAC), una classe di enzimi modificatori epigenomici che sono regolati da fattori sia endogeni che esogeni.
Studi recenti hanno suggerito che l’espressione dell’HDAC dell’ospite è importante per regolare la comunicazione tra il microbiota intestinale e le cellule ospiti.
L’elemento ripetitivo all’interno della cromatina, chiamato nucleosoma, è composto da 147 coppie di basi (bp) di DNA strettamente avvolto attorno a un ottagono istonico.
La metilazione degli istoni è un meccanismo di controllo epigenetico cruciale nei mammiferi che può influenzare l’espressione genica regolando l’accessibilità dei fattori di trascrizione, modificare gli istoni ed i processi di trascrizione.
Questa modifica è regolata tramite l’aggiunta di più componenti e con la rimozione di gruppi metilici.
L’enzima DNA metil-transferasi (DNMT) può aggiungere un gruppo metile dal donatore S-adenosilmetionina (SAM) nell’atomo di carbonio 5 della citosina (5-mC), mentre un componente della famiglia degli enzimi (TET) diossigenasi può invertire attivamente questo processo mediante ossidazione da 5-mC a 5-idrossimetilcitosina (5hmC).
La sintesi di 5-hmC è importante per vari processi biologici e può essere regolata da un certo numero di molecole di piccole dimensioni.
Microbiota intestinale e produzione di metaboliti modulatori epigenetici
Il microbiota contribuisce al microambiente intestinale con prodotti dipendenti dalla dieta, come lipidi, aminoacidi, vitamine, acidi grassi a catena corta (SCFA) e prodotti indipendenti dalla dieta, come il lipopolisaccaride (LPS) ed il peptidoglicano.
L’impatto epigenetico dei metaboliti alimentari prodotti dal microbiota intestinale hanno il potenziale per modificare l’epigenoma delle cellule ospiti e, a loro volta, alterare lo sviluppo e la funzione della cellula.
Numerosi studi recenti hanno esaminato che gli SCFA derivati da batteri commensali prodotti attraverso la fermentazione batterica dei carboidrati alimentari nel colon e che possono essere incorporati dalle cellule epiteliali intestinali, attivano i recettori accoppiati alle proteine G (GPCR), come GPR41 e GPR43.
Per valutare l’azione epigenetica del microbiota intestinale umano, in uno studio sono state divise in due gruppi otto donne in gravidanza e si è proceduto al sequenziamento dei metilomi del DNA in relazione al loro microbiota dominante, cioè i Bacteroidetes, i Firmicutes e i Proteobacteria.
Da tale studio è emersa una chiara associazione che dimostra l’azione epigenetica del microbiota.
I geni con promotori diversamente metilati nel gruppo in cui erano dominanti i Firmicutes erano collegati al rischio di malattia, principalmente a malattie cardiovascolari e in particolare quelle dovute ad alterazione del metabolismo lipidico, all’obesità e alla aumentata risposta infiammatoria.
Questo è uno dei primi studi che evidenzia l’associazione dei phyla batterici predominanti nell’intestino con degli schemi di metilazione.
Nell’ultimo decennio, l’azione epigenetica del microbiota ed i prodotti della sua modulazione naturale hanno guadagnato un crescente interesse.
Composti bioattivi provenienti da varie fonti dietetiche, tra cui thè verde, soia, frutta, bacche, vegetali appartenenti alla famiglia delle Crucifere ed altre verdure, cibi integrali, pesce ed altri prodotti, hanno dimostrato di interagire, sia in vitro che in coltura cellulare, con gli enzimi coinvolti nella regolazione genica epigenetica, tra cui le metiltransferasi del DNA, l’istone acetiltransferasi, le deacetilasi e le demetilasi.
Inoltre, molti agenti dietetici hanno dimostrato di alterare l’espressione dei mi-RNA (non-coding RNA con dimensioni di 17-25 nucleotidi).
Il microbiota intestinale ed i suoi metaboliti potrebbero essere importanti mediatori delle interazioni tra la dieta e l’epigenoma, potrebbero influenzare numerosi meccanismi biologici, incluso il segnale di trasduzione mediato da recettori nucleari e fattori di trascrizione come NF-kB, la proliferazione cellulare e il ciclo cellulare, la progressione, la differenziazione cellulare, la riparazione del DNA, l’induzione all’apoptosi, la motilità cellulare, la formazione di metastasi e la senescenza cellulare.
Gli studi in vivo su modelli animali e umani sono ancora limitati.
Le differenze inter-individuali nel microbioma intestinale possono influenzare il rilascio, il metabolismo e la biodisponibilità di agenti dietetici e spiegare il perché della variabilità nella risposta dell’azione epigenetica del microbiota negli studi sull’uomo.
Alcuni metaboliti microbici, incluso l’acido folico, gli acidi fenolici, le urolitine, gli isotiocianati e gli acidi grassi a catena lunga sono stati studiati riguardo alla loro influenza sui meccanismi epigenetici.
Molecole ad azione epigenetica
Folati e Vitamine del gruppo B
Il folato è un micronutriente critico, una vitamina idrosolubile, che può essere ottenuto da alimenti specifici (verdure a foglia verde, asparagi, legumi, noci, verdure crocifere, avocado, papaia, ecc.) o da integratori alimentari.
I folati sono importanti nella generazione di SAM (S-adenosil-metionina), il principale donatore di metile per l’enzima DNA metil-transferasi (DNMT).
I Bifidobacterium e i Lactobacillus, comuni batteri probiotici, sono capaci di produrre vitamine tra cui l’acido folico.
In uno studio pilota, ai volontari sono stati somministrati tre ceppi probiotici di Bifidobacterium e tutti e tre i ceppi hanno determinato un aumento significativo della concentrazione di acido folico nelle feci, suggerendo che Bifidobacterium può generare folato nell’intestino, che può essere utilizzato dall’ospite in grado di influenzare i modelli di metilazione del DNA.
I batteri inducono anche la demetilazione attraverso l’ossidazione da 5-mC a 5-hmC, processo catalizzato da proteine della famiglia TET.
Negli studi sull’uomo, alcuni gruppi di ricerca hanno scoperto che la carica del batterio Fusobacterium nucleatum aumenta in biopsie prelevate da cancro colon-rettale e che questo correli con i meccanismi di metilazione del DNA.
La metionina dietetica modella la composizione del microbiota dell’ospite e modula anche il metabolismo batterico generando substrati utili per la sintesi dell’S-adenosil-metionina, necessaria per la metilazione del DNA e per la biosintesi della fosfatidilcolina.
Escherichia coli privata di una fonte esterna di metionina riduce l’efficienza della sintesi di fosfatidilcolina nell’ospite.
La fosfatidilcolina può attivare il recettore nucleare NR5A, che sopprime l’espressione del gene grl-21 per inibire la scissione mitocondriale e l’accumulo di lipidi nell’ospite.
Pertanto, l’azione epigenetica del microbiota modifica non solo la dinamica della metilazione del DNA, ma regola anche l’attività dei recettori nucleari.
Oltre al folato, il microbiota intestinale fornisce una varietà di metaboliti energetici alimentari, come l’ATP, il NAD+ e l’acetil-CoA, che vengono utilizzati come cofattori per enzimi di regolazione epigenetica.
Polifenoli
Polifenoli provenienti da varie fonti, quali il thè verde, i lamponi neri, il vino rosso, il caffè, le mele, gli isoflavoni della soia, la curcumina del curry e altri, sono stati segnalati come fattori che influenzano i meccanismi epigenetici in vitro nelle reazioni enzimatiche e nelle colture cellulari.
Isoflavoni
Gli isoflavoni, come la genisteina e la daidzeina, sono composti fitochimici con attività anti-estrogenica e si trovano nella soia e in altri legumi.
Studi epidemiologici suggeriscono che le popolazioni che seguono una dieta asiatica, ricca di prodotti a base di soia, hanno un rischio ridotto di tumori ormono-dipendenti.
L’S-equolo, un composto idrossifenolico, metabolita microbico della daidzeina, ha maggiore biodisponibilità ed azione estrogenica rispetto alla daidzeina.
Gli studi metabolici hanno dimostrato che approssimativamente un terzo della popolazione occidentale e fino al 65% della popolazione asiatica è in grado di produrre S-equol.
Nell’ultimo decennio, sono aumentate le evidenze che gli isoflavoni della soia, incluso il metabolita S-(2) equolo, influenzano l’azione di enzimi epigenetici e modula l’espressione genica per contrastare il cancro.
Catechine
Rispetto alla modulazione dei meccanismi epigenetici, le catechine del tè verde (GTC), come ad esempio l’epigallocatechina-gallato (EGCG) è la principale catechina studiata, appartenente al gruppo dei polifenoli.
In vitro, EGCG ha dimostrato di inibire l’attività e l’espressione degli enzimi DNMT e della demetilazione reprimendo i geni coinvolti nel controllo del ciclo cellulare, nella segnalazione cellulare (RARb), nella riparazione del DNA e nell’apoptosi.
Il metabolismo della catechina da parte del catecol-Omethyltransferase (COMT) porta al consumo di SAM, che è quindi meno disponibile per l’attività catalitica delle metiltransferasi del DNA e istonica.
D’altro canto, le reazioni di metilazione di COMT producono S-adenosil-L-omocisteina (SAH), che è un inibitore del feedback negativo delle metiltransferasi.
Sia l’esaurimento di SAM che livelli elevati di SAH potrebbero causare livelli ridotti di metilazione del DNA o dell’istone, che potrebbe conseguentemente influenzare espressione genica.
Glucosinolati
Le specie vegetali della famiglia delle Crucifere sono una ricca fonte di glucosinolati, precursori di isotiocianati (ITC) e altri composti reattivi con proprietà antimicrobiche e una vasta gamma di attività di prevenzione del cancro, compresa l’inibizione dell’infiammazione e della proliferazione cellulare, nonché dell’induzione dell’arresto del ciclo cellulare, dell’apoptosi e dell’autofagia.
Il rilascio di ITC dai glucosinolati è catalizzato dalla tioglucosidasi di origine microbica e dalla mirosinasi di origine vegetale.
Quando l’attività della mirosinasi viene inattivata dai processi di cottura, questa reazione dipende dalle tioglicosidasi batteriche intestinali.
La diversità del microbioma intestinale può, pertanto, modulare la biodisponibilità degli ITC.
D’altra parte, il consumo regolare di Crucifere può influenzare la composizione delle comunità batteriche intestinali umane.
Nel 2004, alcuni studi hanno rilevato che il sulforafano, il principale ITC rilasciato dai germogli dei broccoli, inibiva l’attività dell’HDAC in vitro.
Quindi, il sulforafano e altri prodotti bioattivi derivanti dalle verdure crucifere influiscono sui meccanismi epigenetici.
Fibra alimentare
La fermentazione della fibra alimentare porta alla formazione di SCFA tra cui acetato, propionato e butirrato.
Il butirrato funge da importante fonte di energia per le cellule epiteliali intestinali, è importante per prevenire la carcinogenesi del colon ed è associato ad effetti anti-infiammatori e antiossidanti, di induzione della differenziazione cellulare, arresto del ciclo cellulare e apoptosi.
L’azione come inibitore dell’HDAC del butirrato fu descritta per la prima volta quasi 40 anni fa.
Molti dei suoi effetti sull’espressione genica e la sua attività antiproliferativa sono correlate ai cambiamenti nella struttura della cromatina.
È stato proposto che il butirrato agisca sul processo di acetilazione dell’istone mediante due meccanismi distinti che causano iperacetilazione dell’istone.
L’inibizione dell’HDAC regola i geni coinvolti nel ciclo cellulare arresto e induzione dell’apoptosi e differenziazione cellulare.
Anche se gli SCFA sembrano essere cruciali per l’azione epigenetica del microbiota sulll’epigenoma dell’ospite, attraverso la metilazione del DNA e le modifiche istoniche, molti dei dettagli del meccanismo rimangono ancora poco chiari.
Considerazioni e conclusioni
L’azione epigenetica del microbiota ha dimostrato di svolgere un ruolo cruciale nella salute e nella malattia.
“Siamo ciò che mangiamo” recita un vecchio aforisma, e molti nutrizionisti sarebbero d’accordo. Lavori recenti evidenziano potenziali interazioni tra microbioma ed epigenoma ospite, che dimostrano un ruolo chiave del microbiota intestinale nella regolazione della risposta dell’ospite nei confronti dei segnali ambientali.
A causa del contatto diretto tra il microbiota e le cellule di vari organi dell’ospite, ma soprattutto con quelle dell’intestino, si ritiene che i metaboliti derivati dall’azione del microbiota siano i meccanismi primari di regolazione dell’epigenoma dell’ospite.
Al momento non è chiaro come i metaboliti batterici agiscano sulle vie di segnalazione che regolano le risposte dell’ospite e quali componenti specifici delle macchine trascrizionali degli ospiti siano modulate per indurre cambiamenti nell’espressione del gene ospite.
Ulteriori studi di decifrazione dei segnali specifici derivati dal microbiota che influenzano l’epigenoma ospite fornirà nuove intuizioni per comprendere nuovi meccanismi che influiscono sulla salute o sulla malattia.
Le scelte dietetiche, a lungo termine, influenzano la diversità e l’espressione genica del microbiota intestinale che fornisce metaboliti energetici cofattori di reazioni epigenetiche. Gli agenti dietetici possono influenzare l’epigenoma direttamente o attraverso il loro microbico metaboliti.
Gli studi futuri devono integrare informazioni sullo stile di vita (assunzione con la dieta, trasformazione degli alimenti, informazioni sul fisico attività, uso di antibiotici, ecc.), genotipo, composizione del microbioma intestinale e metaboloma con informazioni su tutto il genoma sull’epigenoma ed espressione genica/proteica per comprendere appieno, in un approccio di “biologia dei sistemi“, le interazioni all’interno del sistema per influenzare il miglioramento della salute umana.
Sebbene i recenti progressi discussi qui offrano passi promettenti per riconoscere l’importanza dell’epigenomica nel mediare la relazione ospite-microbiota, comprendere i meccanismi e la misura in cui la regolazione epigenomica orchestra questa relazione è ancora agli albori.
Sarà essenziale determinare i percorsi critici e le modalità di azione con cui gli enzimi modificatori epigenomici specifici delle cellule rispondono in modo differenziato ai segnali derivati dai batteri commensali e come regolano l’epigenoma.
Per capire come questi percorsi possano essere manipolati per migliorare le interazioni ospite-microbiota e curare le malattie influenzate dal microbiota richiederanno continue ricerche interdisciplinari di base.
In conclusione, questo obiettivo ambizioso non può che essere raggiunto attraverso progetti di ricerca interdisciplinare, combinando l’esperienza di tecnologi alimentari, nutrizionisti, chimici alimentari, biologi molecolari, epigenetisti, clinici, epidemiologi nutrizionali, bioinformatici e statistici per raggiungere una visione integrata sull’influenza della dieta sulla salute umana.
Fonti
- Chang PV, Hao L, Offermanns S, Medzhitov R. The microbial metabolite butyrate regulates intestinal macrophage function via histone deacetylase inhibition. Proc Natl Acad Sci U S A. 2014; 111:2247–2252.
- Himanshu Kumar, Riikka Lund, Asta Laiho,b Krista Lundelin,a,d Ruth E. Ley,c Erika Isolauri, Seppo Salminen “Gut Microbiota as an Epigenetic Regulator: Pilot Study Based on Whole-Genome Methylation Analysis” MBio ASM. 2014; Volume 5 Issue 6 e02113-14
- Crider, K.S., Yang, T.P., Berry, R.J., and Bailey, L.B. (2012) Folate and DNA methylation: a review of molecular mechanisms and the evidence for folate’s role. Adv. Nutr. 3, 21_38
- Jones P.A. “Functions of DNA methylation: islands, start sites, gene bodies and beyond”. 2012; Nat. Rev. Genet. 13, 484–492.
- Richmond T.J., Davey C.A. “The structure of DNA in the nucleosome core”. Nature. 2003; 423:145–150.