Per “splicing” si intende un processo molecolare che avviene all’interno delle cellule in modo da rendere maturo l’RNA messaggero nascente (mRNA). Questo processo è caratteristico delle cellule eucariotiche (assente in batteri e fagi) in cui i processi di maturazione dell’mRNA prevedono varie fasi. Durante lo splicing infatti, mediante “macchinari molecolari” vengono tagliati gli introni (zone non codificanti del genoma) al fine di dare un risultante costrutto formato da soli esoni, sequenze codificanti. Ormai da tempo è noto che a partire da un gene, si ottengono proteine diverse (da qui il termine “alternativo“), grazie al processo di splicing alternativo. In questo modo cellule di distretti diversi, pur avendo lo stesso genoma, codificano per funzioni biologiche differenti! Infatti, in base alla composizione di esoni (sequenze codificanti) all’interno dell’mRNA, la cellula avrà un “carattere” che le conferisce la capacità di svolgere una funzione piuttosto che un’altra.
Lo splicing
Per comprendere al meglio, partiamo dal concetto di splicing. Lo splicing è un processo necessario all’mRNA appena prodotto in quanto consente di rimuovere alcune parti non codificanti, ossia gli introni, legando tra loro quelle codificanti, cioè gli esoni. Introni ed esoni sono sequenze alternate nel pre-mRNA (RNA messaggero non ancora maturo). Inoltre gli esoni spesso sono sottoposti ad un processo di rimescolamento che permette loro di riposizionarsi (talvolta portando con sè pezzi di introni) all’interno dei geni, creandone di nuovi. Lo splicing è considerato una delle modifiche post-trascrizionali in quanto appunto l’mRNA nascente deve essere rielaborato prima che sia del tutto pronto ad essere tradotto in proteina. Il fatto di avere uno “splicing alternativo” ci permette di produrre proteine differenti a partire da uno stesso gene e questo è dovuto alla modalità di riarrangiamento di esoni ed introni durante il processo di splicing.
Lo spliceosoma, il “macchinario molecolare”
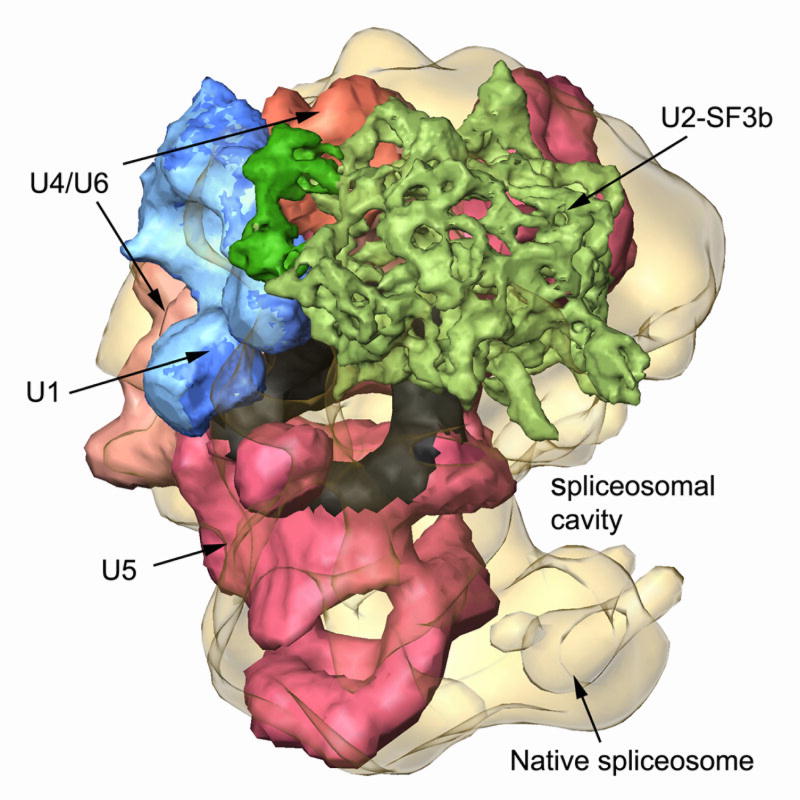
Il cosiddetto “macchinario molecolare”, lo spliceosoma (Figura 1) è il responsabile di due processi distinti e conseguenti: la trascrizione del DNA ad mRNA e lo splicing (anche alternativo). Il primo dei due step è appunto la trascrizione, attraverso la quale a partire dalla molecola di DNA viene reperita l’informazione sotto forma di mRNA (Figura 2). Il 60% dei geni umani è processato mediante splicing alternativo. L’mRNA appena trascritto, come menzionato precedentemente, viene considerato “pre-mRNA” proprio per il fatto che necessita di una sorta di elaborazione dell’informazione in esso contenuta.
![Figura 2: Dal DNA alle proteine, passando per l'mRNA. Il processo di splicing alternativo che ci permette di ottenere un costrutto completo al fine di produrre le giuste proteine.[Fonte:https://www.infors-ht.com/es/blog/mrna-technology-how-a-platform-is-revolutionizing-the-biopharmaceutical-industry/]](https://www.microbiologiaitalia.it/wp-content/uploads/2023/07/mRNA-sintesi.jpeg)
Gli esoni sono le strutture responsabili dell’informazione codificante e perciò dovranno, mediante lo splicing alternativo, essere riuniti tra loro e formare una catena libera da introni (Fig.3). Questo processo avviene all’interno del nucleo, dunque prima che l’mRNA arrivi al citoplasma. Questo meccanismo è finemente regolato da alcune proteine in grado di condurre il macchinario molecolare nei siti appositi al fine di tagliare precisamente nei margini delineanti gli introni senza perciò intaccare gli esoni. Questo arduo compito è svolto dalle proteine “small nuclear ribonuclearprotein” (snRNP), piccole molecole di acido ribonucleico in grado di portare a maturazione l’mRNA.
![Figura 3: Processo di rimozione degli introni dal pre-mRNA (splicing alternativo) , al fine di legare gli esoni tra loro e produrre così il costrutto codificante per le varie isoforme proteiche [Fonte:https://digimparoreda.capitello.it/app/books/CPAC12_8361358A/html/17]](https://www.microbiologiaitalia.it/wp-content/uploads/2023/07/esoni-introni-1024x705.jpg)
I tipi di splicing alternativo
Adesso che conosciamo i protagonisti, cerchiamo di capire il funzionamento nel dettaglio di questo importante processo molecolare. Innanzitutto il processo di splicing alternativo può essere sia costitutivo che regolato. “Costitutivo” rappresenta la condizione per cui un determinato costrutto è sempre prodotto, costantemente, a partire dal gene trascritto. “Regolato” vuol dire invece che un determinato prodotto è generato quando è necessario e quindi in condizioni e distretti differenti a seconda della richiesta cellulare.
Le fasi dello splicing alternativo
Il processo di splicing alternativo ha inizio con una fase definita “Early”. In questa fase una delle proteine snRNP, U1, si lega in posizione 5′ e la proteina BBP (branch point binding protein) riconosce l’adenina nel punto di ramificazione e vi si lega. Successivamente la proteina BBP è sostituita da U2 che richiama dunque il complesso B (formato da U6, U5, U4) permettendo così l’avvicinamento dei due esoni da legare. A seguito di questo avvicinamento, si genera il complesso C in cui U1 è sostituita da U6 provocando così una rottura del legame, allontanando U4. L’unione dei due esoni si completa grazie alla rottura del secondo legame fosfodiesterico mediante U5, liberando così l’introne che era interposto tra gli esoni ora congiunti.
Particolarità dello splicing alternativo
Può capitare che mutazioni della sequenza nucleotidica del DNA possano dare poi prodotti di splicing non funzionanti e originare così delle patologie. Tra le patologie più frequenti derivanti da questi errori annoverino le malattie neurodegenerative. Inoltre le proteine che regolano finemente lo splicing alternativo non sono sempre attivate in quanto potrebbero legare siti detti di silenziamento. Questi particolari siti impediscono la formazione o il reclutamento del macchinario di inizio, annullando così il proseguimento della reazione. Ci sono invece condizioni in cui le proteine si legano a siti “enhancer” capaci di incrementare lo splicing. Questi meccanismi opposti sono indice di come la cellula sia in grado di regolare le proprie funzioni in base alle sue necessità, aumentando o reprimendo la produzione di una proteina!
La funzione degli introni nell’evoluzione
Perché nel genoma sono conservate forme non codificanti? Per molto tempo gli introni sono stati considerati inutili ma non è del tutto vero! Spesso contengono dei miRNA, piccole molecole di RNA attive nella regolazione dell’espressione genica. I miRNA sono inglobati nel “complesso di silenziamento indotto da RNA” (RISC) responsabile del silenziamento genico. Più il sistema è complesso più c’è variabilità e presenza di introni nel genoma. Secondo alcuni gli introni potrebbero essere residui genetici lasciati da alcuni virus nel corso dell’evoluzione. Gli esoni attualmente sembrano essere presenti nel nostro genoma in quantità nettamente inferiore rispetto agli introni (solo un 2% in confronto al 25% di introni). Alcuni geni contengono molte sequenze necessarie allo sviluppo proprio all’interno degli introni stessi. Pertanto sembra che dalla considerazione di “DNA spazzatura” ora sono considerati fondamentali nel caratterizzare gli organismi. Questo permette di dare, condividendo le informazioni, un carattere evolutivo comune nel corso del tempo.
Fonti
- http://goldbook.iupac.org/M03857.html
- https://www.sciencedirect.com/topics/neuroscience/snrnp
- https://oadoi.org/10.1038/nature02871
- https://oadoi.org/10.1016/j.cell.2005.10.022
Crediti immagini
- Immagine in evidenza: https://www.sissa.it/sites/default/files/news/photo5942812575664288195%281%29%202.jpg
- Figura 1: https://europepmc.org/articles/PMC5546952/figure/F9/
- Figura 2: https://www.infors-ht.com/es/blog/mrna-technology-how-a-platform-is-revolutionizing-the-biopharmaceutical-industry/
- Figura 3: https://digimparoreda.capitello.it/app/books/CPAC12_8361358A/html/17]