La tassonomia microbica
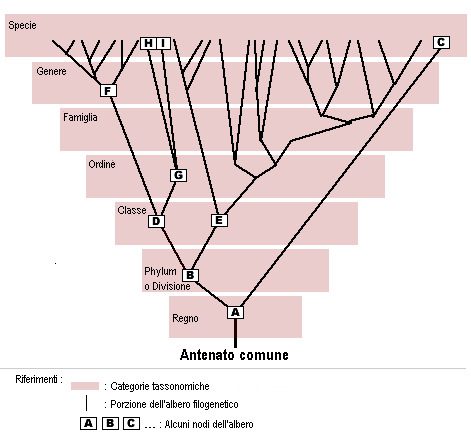
Quando si parla di classificazione filogenetica ci si riferisce al nome scientifico con il quale si indica uno specifico organismo. Per riuscire ad ottenere un ordinamento corretto vengono quindi definite categorie con una specificità via via maggiore. In ordine si hanno regno, phylum, classe, ordine, famiglia, genere e, infine, specie.
La tassonomia microbica comprende l’identificazione degli isolati in specie note, la classificazione di nuovi isolati (creazione di nuovi taxa) e anche la nomenclatura. Mentre le regole della nomenclatura sono stabilite dal Codice di nomenclatura dei batteri, gli schemi tassonomici utilizzati per l’identificazione e la classificazione devono essere affidabili, riproducibili e informativi. È inoltre auspicabile che gli schemi siano facili e accessibili per gli utenti finali della tassonomia.
Nella prima metà del secolo scorso la classificazione era basata sul fenotipo, ad esempio la forma della colonia e gli aspetti fisiologici. Questo metodo determinava la presenza di moltissime nuove specie, per questo motivo si è poi preferito un approccio polifasico. Al fine di determinare la posizione filogenetica di un isolato per valutare le differenze con organismi già caratterizzati e per la descrizione di nuove specie e generi, gli schemi tassonomici si basano ancora oggi sulla classificazione polifasica basata su due diversi approcci:
- La genetica, basata sulle somiglianze tra i microrganismi che possono essere derivate da metodi tassonomici sulla base di caratteri morfologici e fisiologici convenzionali e a marcatori chemiotassonomici come i profili proteici delle cellule intere, il contenuto di % G+C e le omologie DNA-DNA.
- La filogenetica, ovvero il processo di ricostruzione delle possibili relazioni evolutive, che utilizza sequenze nucleotidiche di geni conservati che agiscono come cronometri molecolari.
Gli alberi filogenetici
L’analisi dei geni conservati dell’RNA ribosomiale a piccola unità (rDNA), porta alla costruzione di alberi ramificati che rappresentano la distanza di divergenza da un antenato comune. Essa perciò ha costituito il pilastro della filogenetica microbica. L’approccio presenta però alcune limitazioni, in particolare nella discriminazione di taxa strettamente correlati, e vi è un crescente interesse nell’uso di loci alternativi che differiscono negli individui appartenenti alla stessa specie. Per questo motivo, il confronto del grado di congruenza tra alberi filogenetici derivati da geni diversi fornisce un valido strumento per verificare in che misura essi rappresentino alberi di specie. Inoltre, la rapida espansione delle sequenze genomiche fornirà una ricca fonte di dati per le future analisi tassonomiche, che dovranno tenere conto della struttura di popolazione dei taxa e dei nuovi metodi di analisi delle popolazioni batteriche non clonali.
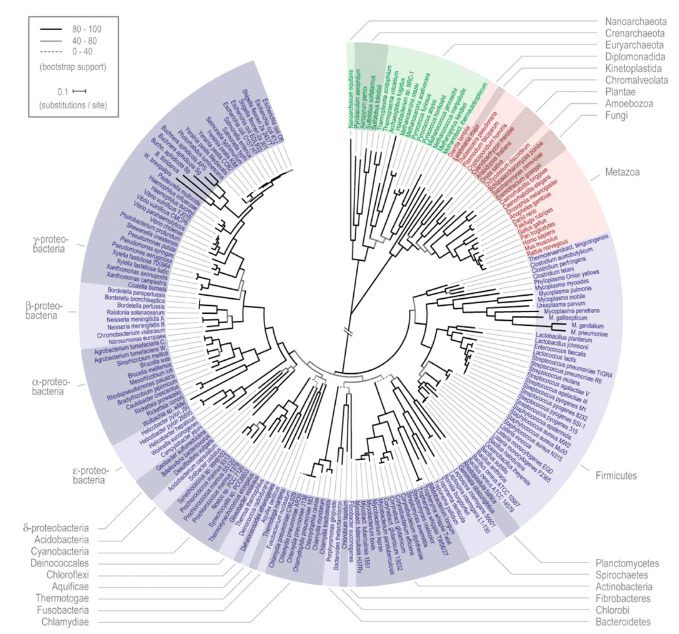
Classificazione filogenetica di specie
La definizione di specie batterica è pragmatica e operativa e mira a stabilire un quadro di classificazione rapido, affidabile, riproducibile e utile basato sull’evoluzione microbica. Nell’ambito della caratterizzazione polifasica, i ceppi della stessa specie presentano fenotipi simili (ad esempio, l’espressione di diversi tipi di enzimi, la capacità di utilizzare diverse fonti di energia e la crescita a diverse temperature e concentrazioni di soluti) e similarità anche a livello fenotipico, formando gruppi ristretti distinguibili. Idealmente, questi gruppi dovrebbero essere facilmente identificabili e distinguibili da specie strettamente correlate.
La nascita del sequenziamento dell’intero genoma (WGS) ha permesso di stabilire una classificazione filogenetica basata sulle informazioni evolutive contenute nelle sequenze genomiche. Inoltre, le attuali tecnologie di sequenziamento sono diventate accessibili per essere utilizzate nell’identificazione microbica di routine. Infatti la tassonomia microbica dipenderà sempre più da strumenti bioinformatici mediante l’utilizzo di sequenze genomiche piuttosto che affidarsi alla caratterizzazione polifasica, che dipende da metodi coltura dipendenti.
Fonti
- Thompson, C.C., Chimetto, L., Edwards, R.A. et al. Microbial genomic taxonomy. BMC Genomics 14, 913 (2013). https://doi.org/10.1186/1471-2164-14-913
- Lapage SP, Sneath PHA, Lessel EF, Skerman VBD, Seeliger HPR, Clark WA, eds. International Code of Nomenclature of Bacteria: Bacteriological Code, 1990 Revision. Washington (DC): ASM Press; 1992.
- Owen, R.J. (2004). Bacterial Taxonomics. In: Woodford, N., Johnson, A.P. (eds) Genomics, Proteomics, and Clinical Bacteriology. Methods in Molecular Biology™, vol 266. Humana Press. https://doi.org/10.1385/1-59259-763-7:353
Crediti immagini
- Immagine in evidenza: https://itol.embl.de/itol.cgi
- Immagine 1: https://it.wikipedia.org/wiki/File:Tassonomia-e-filogenesi.gif
- Immagine 2: https://itol.embl.de/itol.cgi