L’estrazione e la purificazione dell’RNA insieme rappresentano uno step alla base di molte tecniche di biologia molecolare inclusa la RT-PCR (PCR con retrotrascrizione), la preparazione di librerie di cDNA, di microarray e l’analisi attraverso il Northern blotting. Sebbene l’RNA e il DNA presentino caratteristiche diverse, i metodi utilizzati per l’RNA sono molto simili a quelli descritti nel nostro articolo sull’estrazione e purificazione del DNA. In questo articolo vediamo quali sono i punti critici per questa procedura e le metodiche più utilizzate.

Da dove si estrae l’RNA?
Normalmente una cellula di mammifero contiene circa 10-5 μg di RNA, dei quali gli RNA ribosomiali (rRNA) comprendono circa l’80%, gli RNA transfer (tRNA) insieme ad altri piccoli RNA (es. snRNA, snoRNA, scRNA) il 15% e gli mRNA dall’1 al 5%. Quando si parla di estrazione e purificazione di RNA da un campione biologico, generalmente si intende l’RNA totale. I campioni biologici da cui si può estrarre e purificare l’RNA possono essere diversi:
- Fluidi corporei: sangue intero, siero, plasma, espettorato bronchiale, liquido amniotico, liquido seminale, liquido cefalo-rachidiano, urine, etc
- Campioni tissutali (biopsie)
- Cellule in coltura
Affinchè il risultato analitico dell’estrazione e della purificazione di RNA sia preciso, è necessario mantenere le caratteristiche chimiche e biologiche del campione stesso. La qualità più elevata di RNA si ottiene da materiale fresco. Se ciò non è possibile, i campioni prima dell’estrazione devono essere conservati a – 20 / – 80 °C anche per brevi periodi.
La battaglia contro le RNasi
L’estrazione di grandi quantità di RNA è molto spesso soggetta a fallimenti a causa non solo della natura labile dell’RNA, ma anche della presenza ubiquitaria delle ribonucleasi (RNasi). Si tratta di nucleasi che catalizzano l’idrolisi dell’RNA che viene così scisso in componenti più piccole, quindi degradato. Le RNasi sono presenti naturalmente nelle cellule e rilasciate in seguito alla lisi cellulare, uno step essenziale nell’estrazione dell’RNA. Ma perché sono così temute? A differenza delle DNasi, le RNasi non hanno bisogno di cofattori, resistono a trattamenti drastici (ebollizione prolungata, sterilizzazione in autoclave) e sono attive entro un ampio range di pH. Inoltre, anche l’operatore, l’ambiente di lavoro e le soluzioni utilizzate possono essere fonte di RNasi.
E’ importante quindi :
- Dividere tutte le soluzioni utilizzate per l’estrazione e la purificazione di RNA in aliquote, poiché l’uso ripetuto aumenta la probabilità di contaminazioni da parte di batteri che possono rilasciare i loro enzimi, tra cui le RNasi
- Utilizzare la plastica monouso garantita RNasi-free
- Trattare la vetreria per almeno 4 ore a 200°C
- Indossare sempre i guanti e cambiarli spesso, poiché anche la pelle può essere fonte di RNasi
- Mantenere un’area di lavoro separata, dedicata all’RNA, con dotazione di pipette, puntali, provette, reagenti e tamponi esclusivi, per evitare la contaminazione da RNasi ambientali
Inattivazione delle RNasi
Alcune sostanze permettono l’inattivazione delle RNasi durante l’estrazione e la purificazione dell’RNA.Tra queste:
- sali di guanidinio come la guanidina isotiocianato; si tratta di un agente caotropico, cioè un composto organico in grado di scindere i legami idrogeno e i legami idrofobici tra gli acidi nucleici e le proteine. Ad alte concentrazioni (4M) dissolve le proteine facendo perdere la loro struttura secondaria e l’attività biologica.
- 2-mercaptoetanolo, un agente riducente in grado di rompere i ponti disolfuro delle proteine
- Sostanze deproteineizzanti: fenolo, cloroformio, SDS, proteinasi K
- RNase inhibitor: proteine di origine umana (estratte dalla placenta) o non umana (ricombinante), che si legano in modo non covalente alle RNasi e ne inibiscono così più del 90% dell’attività. Il legame è però reversibile e l’inibitore deve essere mantenuto attivo, evitando soluzioni contenenti agenti fortemente denaturanti, come SDS o urea, mantenendo un ambiente riducente (presenza di DTT) e la temperatura <65°C.
- DEPC (dietilpirocarbonato): un agente alchilante molto reattivo che reagisce con le proteine attaccando gli azoti imidazolici dei residui istidinici, determinando la perdita dell’attività enzimatica.
Estrazione organica
L’estrazione organica degli acidi nucleici anche nota come estrazione mediante Fenolo-Cloroformio (in fase liquida) è la metodica più comunemente utilizzata per l’estrazione e la purificazione dell’RNA. Gli step per questo tipo di estrazione sono:
- Lisi cellulare: le molecole di RNA sono relativamente corte e quindi meno facilmente danneggiabili dalle forze frizionali rispetto a quelle di DNA, quindi per la lisi si può far ricorso a metodi vigorosi. La lisi dei tessuti o delle cellule può essere di tipo meccanico, enzimatico o chimico. Tuttavia, deve essere effettuata sempre in presenza di reagenti trattati con gli inibitori delle RNasi, descritti precedentemente. In aggiunta a ciò, durante questa fase è necessario tenere i campioni a 4°C, poichè le RNasi hanno un optimum di funzionamento alla temperatura dell’organismo.
- Purificazione: la fase di purificazione avviene tramite il trattamento con fenolo-cloroformio a pH acido (aggiungendo al composto sodio acetato a pH 4). La miscela è aggiunta al campione che è poi sottoposto a centrifugazione. Fenolo e cloroformio sono immiscibili in acqua, quindi dopo la centrifugazione si otterranno tre fasi distinte della soluzione: una fase inferiore detta organica (fenolica), un’interfase proteica e una fase superiore (acquosa). Il fenolo è un forte denaturante delle proteine, in quanto si lega ad esse mediante legami o ponte d’idrogeno, alterandone la struttura. Le proteine denaturate, con i gruppi idrofobici esposti, diventano solubili nella fase organica precipitando all’interfase fenolo-acqua. Il Cloroformio completa la denaturazione delle proteine, rimuove i lipidi grazie alla sua elevata densità e facilita la separazione della fase acquosa da quella fenolica stabilizzando l’interfaccia tra le due fasi. A pH acido il DNA passa nella fase fenolica, mentre l’RNA rimane nella fase acquosa.
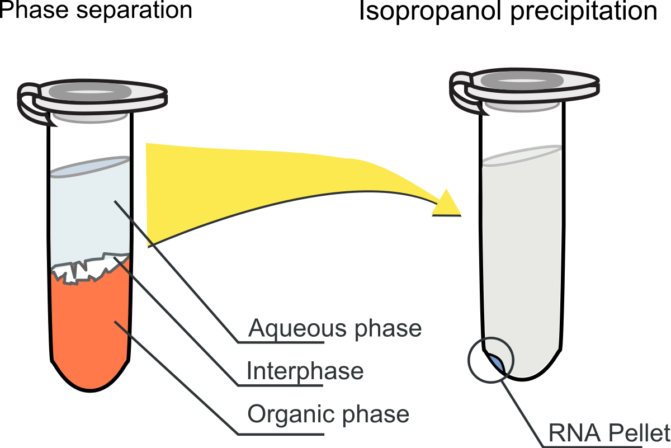
- Precipitazione: lo scopo di questa fase è di concentrare, desalificare e recuperare l’RNA dalla fase acquosa in forma solida. Durante questa fase, si aggiunge un alcol, in particolare l’isopropanolo come soluzione fredda per far precipitare l’RNA. Il pellet è poi lavato in etanolo al 70% e ricentrifugato per eliminare i sali precipitati.
- Risospensione: dopo l’essicazione, il pellet di RNA è risospeso con H2O o con un tampone a bassa forza ionica. L’RNA risospeso deve essere conservato a -80°C.

Estrazione mediante colonnine in spin
Questa è una tecnica estrattiva detta “in fase solida”. Consiste nel legare ed isolare l’RNA in colonnine in spin conteneti una membrana in silice o fibre di vetro. I campioni vengono lisati in una soluzione tamponata contenente inibitori delle RNasi e un’alta concentrazione di sale caotropico. I lisati vengono fatti passare attraverso la colonnina usando la forza centrifuga e l’RNA si attacca alla matrice.
Si effettua un lavaggio in etanolo al 70% per eliminare le proteine residue e i sali. Infine l’RNA è eluito con acqua priva di RNasi.

Metodo di estrazione con sfere magnetiche
Questa metodica per l’estrazione e la purificazione di RNA impiega delle sfere con un nucleo paramagnetico, cioè che hanno proprietà magnetiche solo in prossimità di un campo magnetico esterno. Queste sfere sono rivestite solitamente da una matrice di silice in grado di legare gli acidi nucleici. Per estrarre l’RNA con questo metodo, le cellule sono lisate in un tampone con inibitori delle RNasi e poi incubate con le sfere magnetiche in particolari colonnine, affinchè avvenga il legame tra la matrice in silice e l’RNA.
In presenza di un campo magnetico esterno, posto al di sotto delle colonnine, le sfere sono trattenute sul fondo delle colonnine e il surnatante, contentente proteine e scarti, è rimosso. Successivamente il campo magnetico viene interrotto e l’RNA così isolato è sottoposto ad una serie di lavaggi e infine è eluito dalle sfere magnetiche con acqua priva di RNasi.
Isolamento dell’mRNA
In molti casi è conveniente isolare dalla miscela di RNA totale, l’mRNA eucariotico, che costituisce solo il 2-5% dell’RNA cellulare; a tale scopo si può eseguire una cromatografia di affinità su colonne di oligo (dT) (corti polimeri di desossitimidina) e cellulosa. La maggior parte degli mRNA eucariotici infatti presenta la coda di poliA all’estremità 3’.
La preparazione di RNA totale è fatta passare attraverso la colonna. A elevate concentrazioni saline l’mRNA poliadenilato si ibrida alle sequenze sintetiche di oligo (dT) e viene dunque trattenuto. Al contrario, le altre specie di RNA, che non presentano la coda di poliA, sono eliminate mediante lavaggi con tamponi a bassa concentrazione salina. Infine, l’mRNA adeso può essere eluito con una soluzione a bassa concentrazione salina. Questo metodo non è adatto per gli mRNA batterici, perché solo una piccola percentuale degli mRNA presenta code di poliA piuttosto corte.
Quantificazione
La metodica di quantificazione più comunemente utilizzata in seguito all’estrazione e alla purificazione dell’RNA si basa sull’utilizzo della spettrometria UV (spettro ultravioletto). Gli anelli delle basi puriniche e pirimidiniche che compongono l’RNA sono composti da un’alternanza di singoli o doppi legami. Questi anelli assorbono nell’UV. Ognuna delle quattro basi nucleotidiche assume uno spettro di assorbimento diverso, e lo spettro dell’RNA è la media di questi. In particolare l’RNA ha il suo massimo assorbimento ad una lunghezza d’onda di 260 nm. Secondo la legge di Lambert-Beer, è possibile correlare la quantità di radiazione elettromagnetica assorbita da un mezzo alla concentrazione e allo spessore del mezzo attraversato da essa. A partire da questa legge, possiamo dire che, se l’assorbanza è uguale ad 1 OD (Optical density), saranno presenti in soluzione 40 μg/ml di RNA.

Controllo qualità
Quando si fa l’analisi allo spettrofotometro, viene effettuata anche una lettura a 280 nm e a 230 nm. In particolare, 280 nm rappresenta la lunghezza d’onda corrispondente al massimo assorbimento delle proteine. Il rapporta tra l’assorbanza a 260 nm e quella a 280 nm (A260/A280) indica la purezza dell’RNA. L’RNA è puro se tale rapporto è compreso tra 1.8 e 2.0. E’ importante ricordare che la valutazione della purezza dell’RNA va effettuata in tamponi a pH basico (TE, pH 8.0). L’assorbanza delle proteine presenti nella soluzione, infatti, cambia a pH acidi rendendo imprecisa la determinazione del grado di purezza.
La lettura a 230 nm, invece, riflette la presenza di contaminanti da solventi, quindi da fenolo e carboidrati. In questo caso il valore del rapporto A260/A230 deve aggirarsi tra 2 e 2.2.
Fonti
- https://moodle2.units.it/pluginfile.php/230695/mod_resource/content/1/Estrazione%20di%20acidi%20nucleici.pdf
- K. Wilson e J. Walker. 2001. “Metodologia biochimica. Le bioscienze e le biotecnologie in laboratorio”, V edizione. Cortina Raffaello, ISBN: 9788870786873
- https://lifescience.roche.com/en_it/blog/lab-life/dna-and-rna-purifation/the-top-pros-and-cons-of-different-rna-extraction-methods.html
Fonti immagini
- https://blog.biomall.in/rna-extraction-from-tissue/
- https://www.protocols.io/view/rna-extraction-without-a-kit-beabjaan.html
- https://www.thermofisher.com/it/en/home/life-science/dna-rna-purification-analysis/rna-extraction/rna-types/total-rna-extraction/purelink-rna-mini-kit.html
- https://areteworks.com/work-notables/our-ux-work-nanodrop/01nanodrop-solutionimage/
buongiorno,
per l’utilizzo in preparati in nutrizione clinica (integratori), i nucleotidi dell’RNA da cosa possono essere ricavati?